近年来随着计算机运算能力的提升以及先进算法的涌现,计算机辅助蛋白设计逐渐成为蛋白质工程的研究热点。其中DBTL(design-build-test-learn)循环(图1)是计算机辅助蛋白设计在挖掘新功能酶或改造现有酶的主要思路。
以结构模拟与能量计算为基础的蛋白质计算设计不但能改造酶的底物特异性与热稳定性,还可从头设计具有特定功能的人工酶。在学习和设计模块中,一系列的计算方法,如易于操作的结构预测和分子对接、大规模分子动力学(MD)模拟、高精度量子力学(QM)计算、数据驱动的机器学习和用户友好的网络工具,极大地帮助蛋白质工程改进甚至定制各种功能。
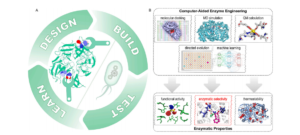
图1 计算辅助加速蛋白质工程
目前应用于蛋白质工程的主要计算工具总结如表1所示。表1列出了目前较为常用的蛋白质结构预测工具、分子对接工具、蛋白质通道计算工具、配体通道工具、分子动力学模拟工具、量子力学与分子力学结合模拟工具、突变靶点确认工具以及可完成多目的Rosetta工具包。其中配体通道工具的是基于分子对接方法预测配体与蛋白之间结合与解结合过程的结构变化,目前应用于蛋白从头设计工程。Gaussian作为QM量化软件,同时支持AMBER等力场,以ONIOM方式计算QM/MM;ORCA同样作为QM量化软件则支持CHARMM力场。另外,QM/MM一项中的chemshell工具是一种接口。当QM量化软件和MM没有相应接口时,如DL-POLY和Gaussian不能同时使用,即想用Gaussian算QM,想用DL-POLY算MM无法实现同时对QM/MM进行计算,chemshell则作为平台出现,可以同时调用上述两个量化软件,支持QM/MM同时进行。
表 1 蛋白质工程的计算工具
| Target property | Tools | Distribution | URL |
| protein structure | MODELLER | SM | https://salilab.org/modeller |
| I-TASSER | WS | http://zhanglab.ccmb.med.umich.edu/I-TASSER | |
| trRosetta | WS | https://yanglab.nankai.edu.cn/trRosetta | |
| molecular docking | AutoDock | SM | http://autodock.scripps.edu |
| UCSF DOCK | SL | http://dock.compbio.ucsf.edu | |
| rDock | SL | http://rdock.sourceforge.net | |
| protein channel | Caver Analyst | SM | http://www.caver.cz |
| CHEXVIS | WS | http://vgl.serc.iisc.ernet.in/chexvis | |
| ligand transport | CaverDock | WS | https://loschmidt.chemi.muni.cz/caverweb |
| MM | GROMACS | SL | http://www.gromacs.org/ |
| AMBER | SL | http://ambermd.org | |
| NAMD | SL | http://www.ks.uiuc.edu/Research/namd | |
| QM and MM | Gaussian | SM | https://gaussian.com |
| ORCA | SL | https://orcaforum.kofo.mpg.de | |
| QM/MM | ChemShell | SL | https://orcaforum.kofo.mpg.de |
| protein design | HotSpot Wizard | SL | https://loschmidt.chemi.muni.cz/hotspotwizard |
| multipurpose | Rosetta | SL | http://www.rosettacommons.org |
表2 用于预测突变效果的工具
| Target property | Tools | URL |
| protein–protein interactions | MutaBind | http://www.ncbi.nlm.nih.gov/projects/mutabind/ |
| iSEE | Be provided as an R-model | |
| mCSM-PPI2 | http://biosig.unimelb.edu.au/mmcsm_ppi | |
| interactions of the protein with nucleic acids and ligands | mCSM-NA | http://structure.bioc.cam.ac.uk/mcsm na. |
| PremPDI | https://lilab.jysw.suda.edu.cn/research/PremPDI/ | |
| mCSM-lig | http://structure.bioc.cam.ac.uk/mcsm_lig | |
| protein dynamics and stability | DynaMut | http://biosig.unimelb.edu.au/dynamut/ |







micronase price – order pioglitazone pill forxiga 10 mg drug
buy clarinex generic – order flixotide nasal sprays oral ventolin
ivermectin for humans – order levaquin pills buy generic cefaclor 500mg
albuterol order – purchase seroflo buy generic theophylline
zithromax 500mg without prescription – buy cheap tinidazole ciprofloxacin usa
cleocin price – buy chloramphenicol medication buy generic chloramphenicol
haRuWXEewV
buy clavulanate no prescription – buy ampicillin pills buy cipro 500mg online cheap
amoxicillin order online – cost trimox 500mg purchase ciprofloxacin generic
clomipramine 50mg over the counter – cymbalta drug doxepin 25mg without prescription
hydroxyzine 25mg tablet – buy buspar 5mg pill order amitriptyline 10mg generic
seroquel 100mg us – buy quetiapine buy generic eskalith online
buy clozaril generic – buy quinapril online famotidine tablet
purchase retrovir sale – order glucophage 500mg allopurinol 300mg usa
order glycomet 1000mg online cheap – duricef 500mg pill lincocin medication
furosemide 100mg over the counter – order candesartan pills buy generic captopril for sale
buy ampicillin generic penicillin usa buy amoxil sale
metronidazole 200mg pill – buy flagyl 200mg pills buy zithromax 250mg online
stromectol buy – cefuroxime online order buy tetracycline 500mg for sale
order valacyclovir online – purchase valacyclovir without prescription brand zovirax 400mg
purchase ciplox generic – generic tindamax order erythromycin 250mg generic
flagyl over the counter – buy generic terramycin for sale zithromax 500mg pills
ciprofloxacin 1000mg price – order ethambutol 1000mg purchase augmentin pill
cipro 1000mg tablet – myambutol brand how to buy clavulanate
buy atorvastatin 80mg pill buy atorvastatin 10mg online cheap oral atorvastatin 40mg