首先,限制性酶切位点就不用多说了吧,它就是DNA分子上被特异性的核苷酸序列(4-8bp),能被限制性内切酶识别,从而切割。
保护碱基:限制性内切酶识别特定的DNA序列,除此之外,酶蛋白还要占据识别位点两边的若干个碱基,这些碱基对内切酶稳定的结合到DNA双链并发挥切割DNA作用是有很大影响的,被称为保护碱基。【百度百科】
Kozak序列:KOZAK是一个女科学家,她研究过起始密码子AUG周边碱基定点突变后对转录和翻译所造成的影响,并总结出在真核生物中,起始密码子两端序列为:—— G/N-C/N-C/N-ANNAUGG——,如GCCACCAUGG、GCCAUGAUGG时,转录和翻译效率最高,特别是-3位的A对翻译效率非常重要。该序列被后人称为Kozak序列,并被应用于表达载体的构建中。
所以,引物设计上游引物就是在起始密码子前面5‘-保护碱基+酶切位点+Kozak+ATG+一小段目的基因序列
下游引物不用加Kozak序列,通常是5‘-保护碱基+酶切位点+终止密码子+(如果你有要表达的标签序列)+一小段目的基因序列
注意,我这里的介绍的引物设计针对目的基因要表达的,pcr出来后是要连接到表达载体上的,所以引物比较长,一般都有40多bp了,不过一样能够p出来。
好了,下面随便找一个基因为例来实战一下,比如我要将Stat5a从一个公司给的质粒上p出来,然后连接到一个表达载体上,采用直接双酶切目的基因和表达载体,然后连接的方式。在UCSC上找到Stat5a的CDs区。
从起始密码子ATG到终止密码子TGA复制到一个word里,
接下来用DNAMAN看这段CDs区里面存在哪些酶切位点,然后根据你表达载体多克隆位点存在哪些酶切位点,选择两个在表达载体多克隆位点有但是CDs区里面没有的两个酶切位点进行设计。不然内切酶把你的目的基因切断了就玩不下去了。。。。。
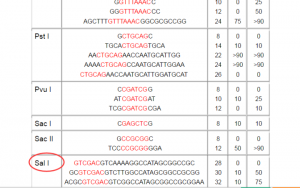
假设我以BamH1、Sal1分别作为上下游的酶切位点
好了,现在去百度找酶切位点保护碱基
百度文库很多这样的表,找到BamH1、Sal1,注意该表格后两列表示酶切2h、20h的酶切效率,当然是选最高的那个最好。
百度文库很多这样的表,找到BamH1、Sal1,注意该表格后两列表示酶切2h、20h的酶切效率,当然是选最高的那个最好。
上游:CGCGGATCCGCCACCATGGCGGGCTGGATTCAGGC
下游(特别注意要你设计出来的要反向互补):GCGTCGACTCAGGACAGGGAGCTTCTA
好了,引物就这样设计好了,很简单有没有~~~
??用DNAMAN怎么找酶切位点,下次再说~~~
—————————————————————————————————————————
致力于简单科普的小白~~~~
辛苦码字,请默默点赞个吧~~~






how to buy micronase – pill forxiga 10mg buy dapagliflozin for sale
cheap ventolin 2mg – advair diskus inhalator price theo-24 Cr without prescription
covid and ivermectin – buy eryc 500mg online generic cefaclor
zithromax 500mg pill – order azithromycin 500mg sale ciprofloxacin 500 mg ca
buy cleocin 300mg online – acticlate brand chloromycetin tablet
order amoxil online cheap – order cefadroxil online cheap cost ciprofloxacin
purchase amoxiclav pills – linezolid oral order cipro 500mg pill
hydroxyzine 10mg oral – cheap atarax 25mg how to buy endep
order anafranil 50mg – buy tofranil order generic sinequan
buy seroquel 50mg generic – eskalith ca how to buy eskalith
clozapine drug – purchase glimepiride generic order generic famotidine
zidovudine without prescription – metformin over the counter purchase allopurinol for sale
glucophage 500mg drug – buy epivir generic order lincomycin 500mg for sale
furosemide ca – prazosin 1mg brand captopril 25 mg cost
order ampicillin pill buy amoxicillin online purchase amoxil generic
order metronidazole 400mg – cefaclor 250mg drug buy generic azithromycin over the counter
ivermectin 6mg pills for humans – purchase suprax online cheap purchase sumycin online
order valtrex 1000mg sale – order valacyclovir online cheap buy zovirax 800mg online
cheap ciprofloxacin – buy doryx pills buy erythromycin 500mg pill
flagyl brand – buy cefaclor cheap buy generic azithromycin over the counter
oral ciprofloxacin 1000mg – keflex 500mg us augmentin 625mg cost
cipro usa – buy augmentin online cheap buy generic augmentin 375mg
atorvastatin 20mg ca cost atorvastatin 10mg how to buy atorvastatin