2021年11月,东京大学Koji Tsuda团队和日本东北大学Mitsuo Umetsu团队共同在ACS Catalysis发表题为“Machine-Learning-Guided Library Design Cycle for Directed Evolution of Enzymes: The Effects of Training Data Composition on Sequence Space Exploration”的文章。
机器学习(ML)正在成为蛋白质工程中的一种有吸引力的工具,在ML引导定向进化过程中,训练数据集中通常不含有“高适应度”突变体,本文作者使用存在“高适应度”突变体5M的分选酶A(SrtA)研究是否包含这样突变体的训练集对序列空间探索的影响。
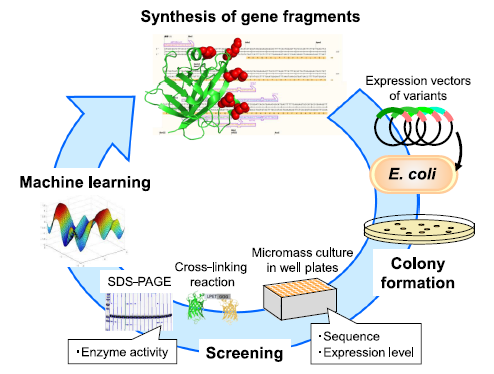
该团队此前提出了一种用于荧光蛋白的ML引导的定向进化方法,作者将该方法拓展到具有迭代设计文库的酶,形成文库设计循环如图1所示。
图1 文库设计循环
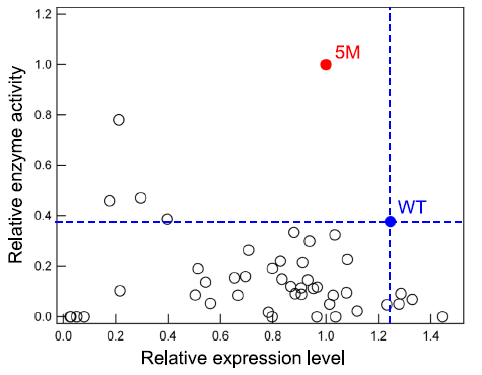
5M突变体具有五个单点突变:P94R、D160N、D165A、K190E和K196T(图2)。文章对这五个位点进行单点突变和组合突变构建了80个突变体初始文库(56个来自所有五个位点的组合突变,24个来自单点突变)。随后实验测量了其中45个突变体的酶活,其余突变体表达水平不足以测量酶活。45个突变体中只有4个显示出比野生型更高的活性,且均低于5M(图3)。
图2 SrtA结构及五个突变残基
图3 突变体的表达水平及酶活
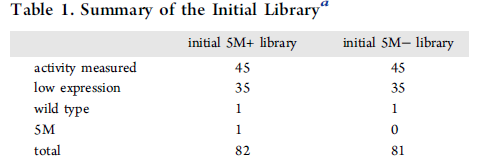
作者基于高斯过程构建了ML模型并进行氨基酸描述符选取的测试实验,发现Z-scale和位置权重矩阵(PSSM)的组合实现了最佳预测精度。为了研究训练集组成的影响,准备了包含80个突变体(将35个表达水平不足的突变体的活性视为0)、野生型及5M的5M+库和不包含5M的5M-库(表1)分别了训练两个ML模型。使用模型对五个突变残基(205=3,200,000个突变体)的所有突变体的活性进行预测并进行排名,排除掉训练集中已有的突变体。在利用5M+数据集训练的模型的预测结果中,之前报道的6个高活性突变体均排在了前2%(表2),利用5M-数据集训练的模型也表现出相当的可靠性。
表1 两个训练集的组成
表2 6个高活性突变体排名情况
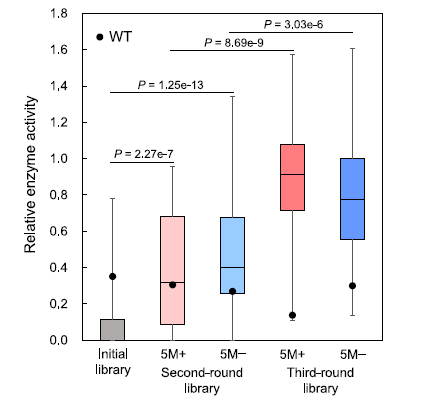
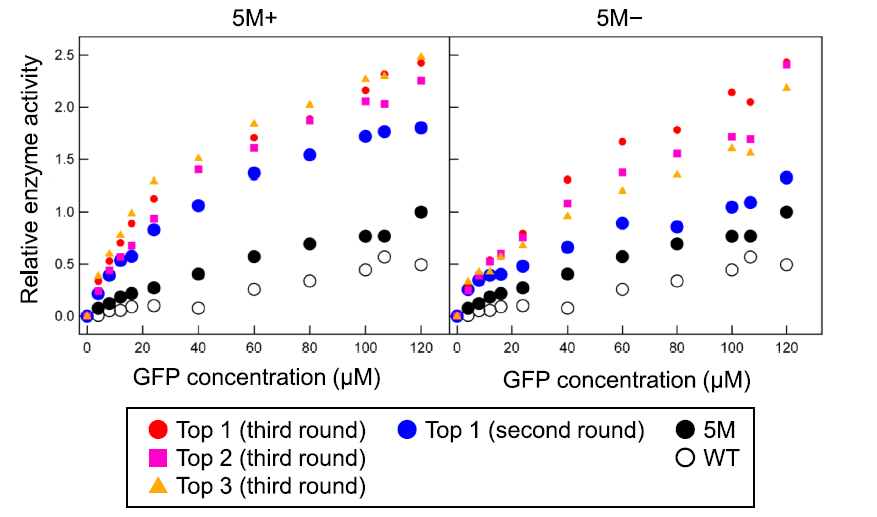
将5M+预测结果排名前50中的37个加上排名51-606之间的30个突变体以及5M-预测结果排名前50中的45个加上排名52-4238之间的31个突变体用于构建第二轮文库。第二轮文库中的许多突变体表现出比野生型更高的活性(图4),第二轮5M-文库中的三个突变体显示出比5M更高的活性,而第二轮5M+文库中的一些突变体也显示出与5M相当的活性。
图4 初始文库、第二轮文库和第三轮文库的酶活
第二轮文库的结果被添加到数据集中,继续用来训练ML模型。然后以同样的方法构建第三轮文库,大多数突变体显示出比野生型更高的活性(图4),其中5M+文库中的19个突变体和5M-文库中的12个突变体的活性高于5M。
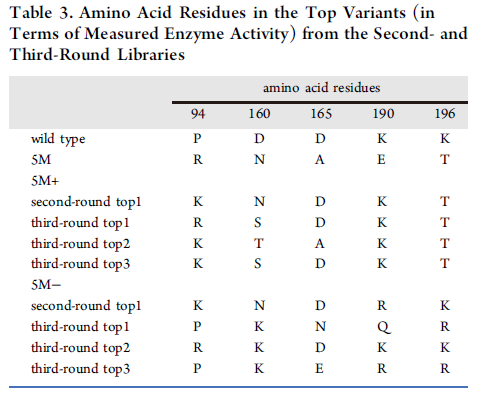
作者选择第二轮文库中最佳的突变体和第三轮文库中排名前三的突变体(表3)进行酶活的详细评估:在不同浓度的底物GFP和单一浓度的底物YFP下测量纯化后突变体的酶活性(图5)。所有突变体均显示出比野生型和5M更高的酶活性,其中来自第三轮文库的突变体分别显示出比野生型和5M高4.4-5.0和2.2-2.5倍的活性。
表3 表现最佳的突变体的氨基酸残基
图5 不同底物浓度下的酶活
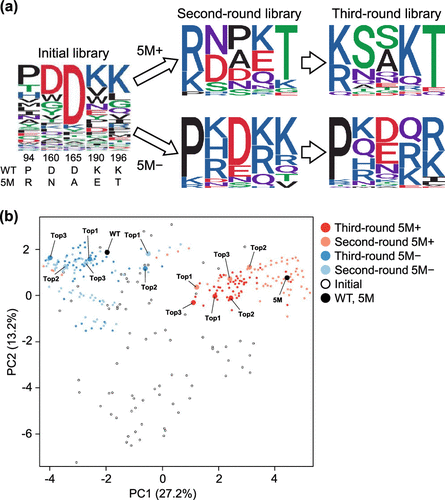
尽管酶活性同样有相当的提升,但突变体的序列在5M+和5M-文库之间存在很大差异(图6a)。为了进一步可视化训练集组成对序列空间探索的影响,作者基于ML模型中使用的序列衍生特征向量对突变体进行了主成分分析(图6b)。在5M+的情况下,ML引导了5M周围的进化,在离5M稍远的位置发现了最高活性的突变体;而在5M-的情况下,ML引导在野生型周围的序列空间进行探索,并最终在野生型周围发现高活性突变体。
图6 序列空间探索的可视化
为了测试这个迭代过程的饱和度,第三轮库的结果被进一步用作ML模型的额外训练数据来预测第四轮库,与第三轮相比,第四轮预测的突变体中酶的活性的最大值没有提高,这意味着迭代在第三轮已经饱和。
为了验证在其他蛋白质中是否也能观察到类似的效果,作者使用GB1数据集进行计算实验,该数据集中含有GB1蛋白中四个位点所有可能突变体的适应度值。同样的,通过构建两个数据集,其中一个包含一个“高适应度”突变体HPV,并分别训练ML模型,证实了HPV的影响类似于在SrtA中观察到的效果。
总之,作者的研究证明了在ML引导的定向进化中调整训练集组成的重要性,强调在训练集中包含“高适应度”突变体对于利用ML进行蛋白质进化取得优异突变体并不总是必要的。此外,使用训练数据子集多次执行ML可能会在序列空间的不同区域内发现优异的突变体。
论文链接:
https://pubs.acs.org/doi/10.1021/acscatal.1c03753?ref=PDF







where can i buy micronase – glucotrol 5mg us pill dapagliflozin 10 mg
clarinex 5mg canada – order triamcinolone 10mg for sale buy albuterol pill
albuterol pills – purchase seroflo inhalator for sale theophylline drug
ivermectin 12 mg oral – ivermectin 6 mg without a doctor prescription cost cefaclor
amoxicillin pills – order cephalexin online cheap cipro sale
clavulanate canada – buy ethambutol generic brand ciprofloxacin 1000mg
order hydroxyzine pills – cheap buspar 10mg endep oral
buy anafranil 25mg pills – buy cheap aripiprazole purchase doxepin pills
order seroquel 50mg pills – where can i buy seroquel eskalith where to buy
purchase clozapine generic – pepcid cheap order pepcid 40mg online cheap
retrovir 300 mg uk – order zyloprim 100mg online zyloprim 100mg sale
buy cheap generic lasix – buy lasix 100mg pill buy capoten 25mg pill
order ampicillin sale purchase monodox pill order amoxicillin generic
flagyl over the counter – cost amoxicillin zithromax 250mg price
ivermectin 12 mg tablets for humans – order sumycin generic order tetracycline 500mg for sale
valacyclovir for sale – buy starlix without prescription zovirax 800mg cost
oXgcvFLGbB
generic ciplox 500mg – trimox pills
erythromycin 500mg over the counter
buy flagyl 200mg – oral flagyl 200mg order zithromax for sale
buy generic ciprofloxacin – order cipro 500mg pills amoxiclav online order
cipro 500mg price – buy keflex for sale buy augmentin 1000mg online
buy lipitor 20mg pill order atorvastatin 10mg without prescription buy atorvastatin 80mg online cheap
GLopxrFQERjSbXP