2021年7月,斯坦福大学的Stephen R. Quake《生物化学杂志》(JOURNAL OF BIOLOGICAL CHEMISTRY,缩写JBC)发表了一篇温度对聚合酶保真性影响的文章。这篇文章为反应温度(生物化学的一个关键参数)如何影响适应各种热环境的生物体的DNA聚合酶保真度提供了有用的参考。
极端微生物的发现有助于开发突破性技术,例如PCR。温度变化通常是这些技术平台的必要步骤,但温度对不同来源聚合酶错误率的影响尚未得到充分研究。
这篇文章分析来自嗜冷、嗜温和嗜热来源的 DNA聚合酶的错误率。研究发现反应温度显著增加了嗜冷和嗜温DNA聚合酶的取代和缺失错误率。基序分析表明,取代错误谱根据聚合酶的系统发育相似性而不是反应温度进行聚类,因此表明反应温度会增加聚合酶的全局错误率,而与序列背景无关。有趣的是,研究还发现嗜冷细菌的DNA聚合酶I在所有温度范围内都比其嗜温同源酶表现出更高的聚合活性,包括低至-19℃,这远低于水的冰点温度。
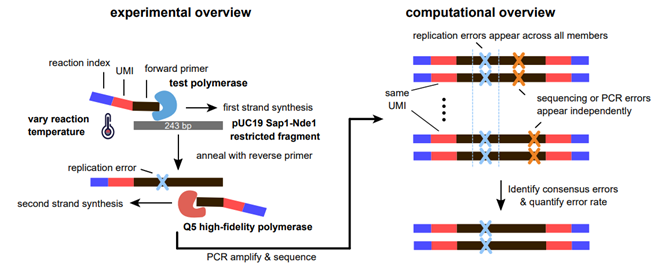
图1 研究的流程和方法
在这一工作中,研究者主要研究流程如图1所示。
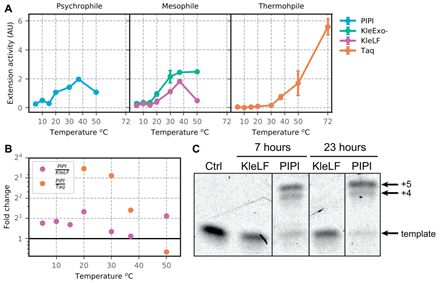
图2 DNA聚合酶在不同温度下的活性表现
研究表明嗜冷来源的DNA聚合酶比嗜温和嗜热聚合酶活性更高,可以在零摄氏度以下复制DNA,如图2A所示。引人注目的是,研究发现PIPI保留了延伸活性(图2C),因为它在7小时内完成了具有5个核苷酸缺口的引物模板的延伸,而KleLF则没有。而且KleLF也未能在-19℃至23小时完成延伸,PIPI用于零下反应的可能性,为新的生化应用打开大门。
图3 高通量多重测序方法计算错配率
然后,研究者又通过高通量多重测序方法(图3),可以将复制错误(由蓝色十字突出显示)与测序错误(由橙色十字突出显示)区分开来。以此证明在不同反应温度下聚合酶错误率。
图4 DNA聚合酶活性-保真度的分析
DNA聚合酶活性-保真度的分析实验表明(图4),反应温度的增加会导致错配比率,而线性回归模型预测结果表明,聚合酶活性每增加一倍,聚合酶错误率就会增加约2.2倍。
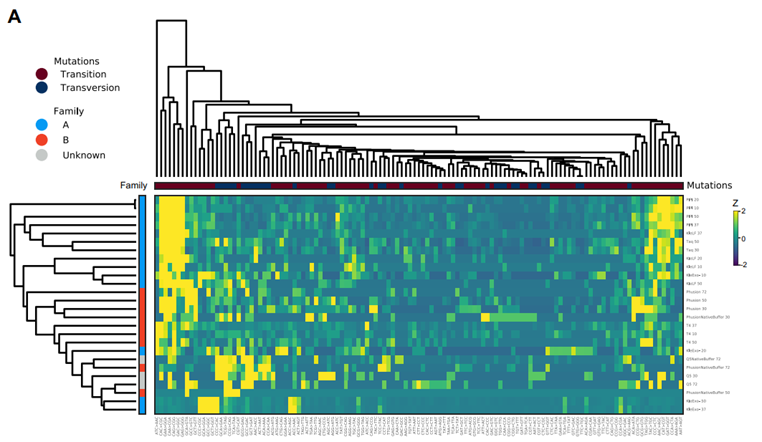
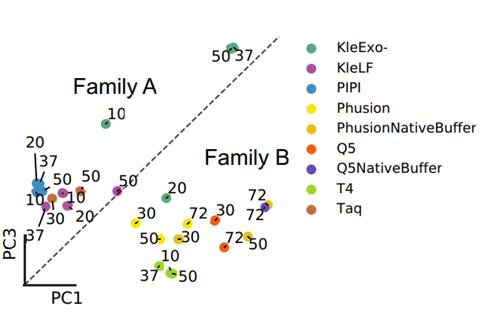
图5 三碱基聚体取代率的分层聚类图
然后,研究者进行了自然标准化三聚体取代率的分层聚类分析(图5),发现取代谱倾向于通过聚合酶的系统发育距离聚类,其中由家族A和B产生的取代在很大程度上分为不同的簇,这与它们各自的温度适应范围无关。这说明了温度对聚合酶取代错误的影响与前后的序列背景无关,不同的突变谱由聚合酶家族类型驱动。
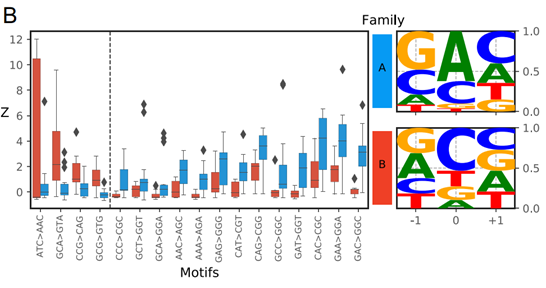
图6 最能区分A家族和B家族错误的替换基序
为了确定最能解释观察到的替代谱的突变谱,研究者训练了一个随机森林决策树来对每个谱的聚合酶家族起源进行分类。通过对每个特征的学习权重进行排序,研究确定了每个聚合酶家族最有区别的突变足迹。如图6所示,最能区分A族和B族聚合酶产生的错误的取代基序。徽标图量化了聚合酶家族A(顶部)或B(底部)生成的-1、0和+1位取代基序的频率。当在-1位置之前是G/C时,表明聚合酶家族A更频繁地产生A→G转变,在-1位点前有嘌呤(A/G)时,聚合酶家族B更频繁地产生嘧啶(C/T) 的颠换。
图7 标准化的3聚体错误率进行了主成分分析
标准化三聚体取代率的主要成分分析表明,较高的主成分1与B家族的替代谱相关,而较高的主成分3与A族的替代谱相关。替代谱投影到主成分1和3上,自然决策边界将A家族和B家族的替代谱分隔开来,这表明两个聚合酶家族之间保守的结构差异可能是聚合酶产生不同突变特征的基础。









micronase 5mg ca – buy micronase pills order forxiga 10 mg pills
cost clarinex 5mg – brand zaditor 1mg order ventolin generic
albuterol inhalator sale – phenergan pills order theophylline 400mg sale
stromectol otc – buy eryc tablets cefaclor 250mg cheap
cleocin 150mg pill – order cefixime 100mg generic order chloromycetin pill
purchase zithromax pills – ofloxacin 400mg cost how to buy ciplox
amoxil pills – buy cefadroxil 500mg pill ciprofloxacin 500mg without prescription
augmentin 625mg canada – buy generic augmentin order baycip online cheap
order hydroxyzine generic – pamelor for sale endep canada
anafranil 25mg cheap – aripiprazole 30mg pills doxepin 75mg price
buy quetiapine 100mg – buy eskalith no prescription cheap eskalith
clozaril 100mg cost – frumil price purchase famotidine sale
zidovudine 300mg over the counter – order epivir pills buy zyloprim pill
order glycomet 1000mg pill – bactrim for sale buy lincocin generic
lasix for sale – order captopril sale captopril 25 mg cheap
purchase ampicillin generic doxycycline buy online amoxil usa
cost valtrex 1000mg – order diltiazem 180mg without prescription buy zovirax 800mg
ivermectin 3mg pills for humans – suprax ca purchase sumycin generic
ciplox brand – cost amoxicillin 500mg
erythromycin 250mg pills
order metronidazole 400mg pills – buy amoxil medication zithromax 250mg usa
order generic ciprofloxacin 500mg – purchase cephalexin generic augmentin 625mg tablet
cipro without prescription – buy cephalexin 125mg without prescription augmentin 1000mg without prescription
buy lipitor 40mg online buy atorvastatin medication lipitor 40mg for sale