编者按
小编以为,合成生物学并不是一个异军突起、凭空创造的领域或学科;却以为合成生物学是,当基因学、蛋白工程学、细胞学等等学科发展并积累到一定程度以后,人们利用各种跨领域知识或技术去解决一些物质生产问题、满足一些生物制造要求,而且渐渐形成的一种高效性、战略化使用自然生物大分子的理念。我们已经知道的是,合成生物学概念形成至今,多数工作被集中在寻找和构建标准化的“基因”及“基因表达”相关的DNA片段上,但我们对合成生物学未来的愿景绝不仅仅于此,离真正形成可编程可调控的一体化细胞工厂,还有很大的距离。小编在本文带大家回忆合成生物学发展到现在的一些大事,分析分析合成生物学到底发展成什么规模了。
我们可以把一个细胞本身想象成一个自动化的流水线工厂,细胞的基因组就是控制这条流水线的电脑。电脑的特点是什么,可以通过接受人为指令执行想要的程序。人们通过对基因的认识,已经全面掌握了“构建基因–表达特定蛋白–形成特定功能”的技术,也就是说现在创造电脑去控制这条流水线的能力已经有了。那么对这条流水线上除基因组以外的其他部分呢?其实也是可以通过重新构建来优化的。
合成生物学启发了我们,从细胞级而不是分子级来看待生物组件可拆分、可重组的特性。我们将模块化的生物组件重新拼在一起,其实可以看成是一个加速地细胞向特定功能进化的过程。相信最终是一定能达到完全模块化细胞组件的高度,虽然这个目标现在看来是十分遥远的。普遍认为合成生物学的发展分为两个阶段【1】,而我们现在正处在第一阶段末和第二阶段初的位置:第一阶段(First Wave)主要集中在发现、构建和优化基因组件;第二阶段(Second Wave)不在仅仅局限于构建基因组件,还要发现其他组件,最终完成更复杂更完善的重组,比如定制细胞。
在合成生物学发展的第一阶段,基本的基因组件,比如启动子(promoter)、核糖体结合位点(RBS)和转录抑制位点(transcriptional repressor),被大量发现,并合理组合成能够调控基因表达的模块。到目前为止,已经发展出来的模块有:开关(Switch)、cascade、pulse generator、延时设置(time-delayed circuit)、振荡器(oscillator)、spatial patterning和logic formulas。但是这些模块并不是死板地拿来用,每次遇到的情况也许不一样,所以每当考虑到构建一个基因模块的时候,基本策略大概有三种:1)重复理性设计(Iterative rational design),尝试设计—实验验证—改观—再验证;2)高通量组合筛选(Varients combination),不管怎么设计,就这几个了,然后再筛选就好;3)定向进化。
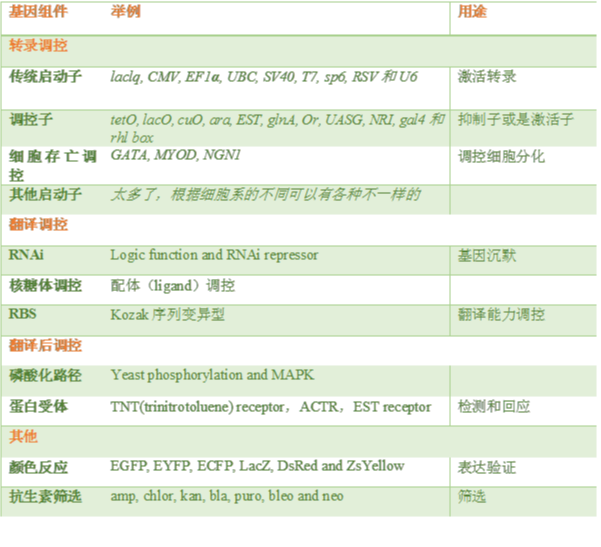
其实构建这些基因模块并不难,难的是我们如何让各个基因组件相互磨合,毕竟现在发现的这些基因组件大部分不是来自同一个生物的基因组的。详情请见下表,基本全部已知的基因组件:
Table 1, 用于构建各色模块的基因组件【1】
第一波合成生物学的终极目标是完成各种标准基因组件入库,也就是说在未来,任何人都可以随意查询到各种功能的标准组件,并且进行随意的混合和组合。在第一波合成生物学的大潮中,iGEM的比赛应运而生,从2004年第一届iGEM开始,来自全世界各个大学的学生们脑洞打开发挥想象组成挑战小组,设计并构建自己的基因组件完成特定功能。查看BioEngX往期文章了解iGEM比赛。
在获得这些简单的基因组件后,科学家们也构建了简单的基因模块,举三个例子吧:
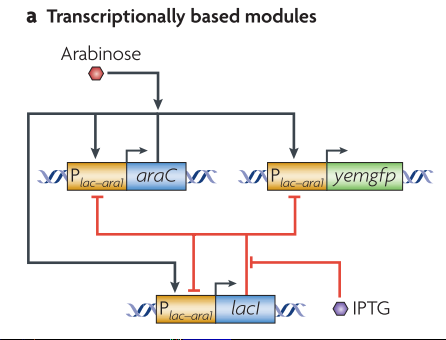
转录调控模块【2】
这个被称为双重回馈震荡模块,首先我们先看图中橙色部分,三个一模一样的启动子,这个启动子是既可以被AraC激活也可以被Lacl抑制;然后我们来看两个蓝色部分,都是转录调控的基因组件,第一个araC用于启动子激活,而lacl用于启动子抑制,AraC和Lacl又分别可被外界配体阿拉伯糖(arabinose)和IPTG结合调控;最后我们看绿色的部分,用于酵母内表达的绿色荧光蛋白。图中黑线箭头代表激活,红线代表抑制阻断,整个系统为了能够稳定表达绿色荧光蛋白,同时我们可以通过调控阿拉伯糖和IPTG的比例来调控荧光的表达量。这个模块是不是很完美,虽然有三段基因参与,但调控速度并不慢。
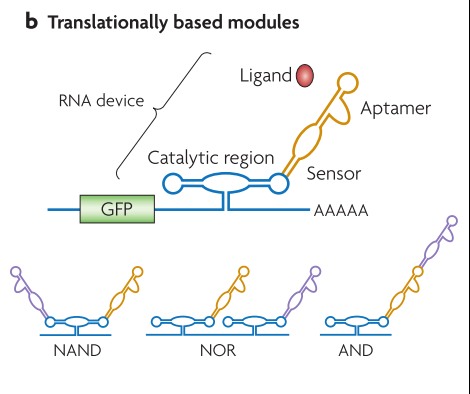
翻译调控组件【3】
首先要说的是图中都是RNA结构,不是DNA。先看棕色的部分,Aptamer其实就是适体,短链功能型RNA。这里aptamer的意义就是在于,当配体ligand和aptamer结合的时候,RNA上的催化区间(catalytic region)的结构会发生变化,要么加速核糖体自身分解亦或是延缓核糖体分解,因此影响到整段coding RNA在细胞中存活的时间,因而调控GFP蛋白的表达。值得一提的是Aptamer和catalytic region的位置和组合有多重多言个,甚至可以引入多个aptamer。
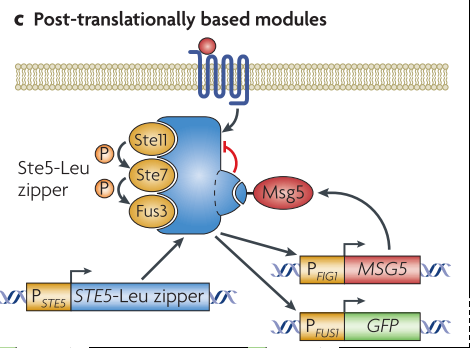
翻译后调控【4】
其实翻译后的调控模块,多是模仿显示细胞存在的一些信号路径,自然界已经帮我们构建好了一些非常高效地回路,我们只需照葫芦画瓢好了。上图是个新建的磷酸化抑制模块,为什么是翻译后调控呢,就是参与调控的蛋本身要先被表达出来,再来调控自身和目标蛋白的表达。最重要的是红色的部分,MSG5用于抑制反馈,从而控制GFP表达。
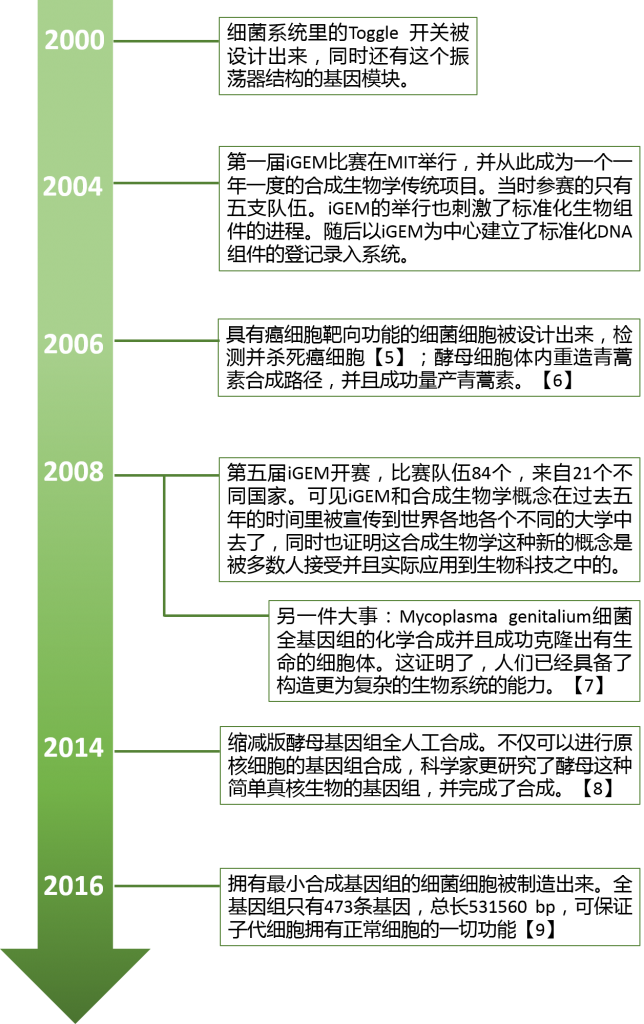
给各位看一份合成生物学发展的时间图吧,如下,
现在已经发展到接近第一波潮流的末端了,正在进行第二波发展,也就是形成更复杂更加高级的系统,比如构造非自然最小基因组、构造非自然最简单细胞。关于这个话题,我们以后再聊。
1, Purnick, P. E. & Weiss, R. (2009). The second wave of synthetic biology: from modules to systems. Nat Rev Mol Cell Biol, 10, 410-22.
2, Stricker, J., Cookson, S., Bennett, M. R., Mather, W. H., Tsimring, L. S., & Hasty, J. (2008). A fast, robust and tunable synthetic gene oscillator. Nature,456(7221), 516-519.
3, Win, M. N., & Smolke, C. D. (2008). Higher-order cellular information processing with synthetic RNA devices. Science, 322(5900), 456-460.
4, Bashor, C. J., Helman, N. C., Yan, S., & Lim, W. A. (2008). Using engineered scaffold interactions to reshape MAP kinase pathway signaling dynamics. Science, 319(5869), 1539-1543.
5, Anderson, J. C., Clarke, E. J., Arkin, A. P., & Voigt, C. A. (2006). Environmentally controlled invasion of cancer cells by engineered bacteria.Journal of molecular biology, 355(4), 619-627.
6, Ro, D. K., Paradise, E. M., Ouellet, M., Fisher, K. J., Newman, K. L., Ndungu, J. M., … & Chang, M. C. (2006). Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 440(7086), 940-943.
7, Gibson, D. G., Benders, G. A., Andrews-Pfannkoch, C., Denisova, E. A., Baden-Tillson, H., Zaveri, J., … & Merryman, C. (2008). Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome.science, 319(5867), 1215-1220.
8, Annaluru, N., Muller, H., Mitchell, L. A., Ramalingam, S., Stracquadanio, G., Richardson, S. M., … & Cai, Y. (2014). Total synthesis of a functional designer eukaryotic chromosome. Science, 344(6179), 55-58
9, Hutchison, C. A., Chuang, R. Y., Noskov, V. N., Assad-Garcia, N., Deerinck, T. J., Ellisman, M. H., … & Pelletier, J. F. (2016). Design and synthesis of a minimal bacterial genome. Science, 351(6280), aad6253.
管理员邮箱:info@bioengx.org;管理员微信:bioengxadmin; 内容由作者原创。欢迎留言讨论哦,如需再转载,请联系管理员。
扫描下方二维码关注BioEngX官方微信公众平台










cost desloratadine – order flixotide online order ventolin 2mg generic
ivermectin 3mg tablet – cheap levofloxacin 250mg generic cefaclor 500mg
purchase ventolin inhalator generic – order theophylline 400 mg online cost theo-24 Cr 400 mg
cleocin 150mg us – where can i buy clindamycin chloromycetin over the counter
buy clavulanate generic – generic ciprofloxacin buy baycip pills for sale
buy amoxicillin pills – ceftin price ciprofloxacin over the counter
anafranil 50mg pills – purchase anafranil for sale sinequan usa
buy hydroxyzine 25mg without prescription – buy atarax sale amitriptyline 10mg uk
cost seroquel 50mg – geodon 40mg cost order eskalith generic
clozaril canada – order ramipril sale buy pepcid 40mg
buy metformin without prescription – purchase cipro without prescription lincocin canada
order zidovudine 300 mg online – order zyloprim 100mg pill
cheap furosemide 40mg – buy coumadin online purchase captopril online
buy acillin tablets buy acillin cheap buy amoxicillin online cheap
order flagyl online – buy amoxil without a prescription buy zithromax medication
ivermectin india – amoxiclav over the counter cost tetracycline 250mg
valacyclovir 1000mg generic – acyclovir cost acyclovir over the counter
ciprofloxacin 500 mg ca – chloramphenicol pills buy erythromycin 250mg
buy metronidazole medication – metronidazole pills order zithromax 500mg
purchase ciprofloxacin online cheap – cephalexin 500mg uk amoxiclav oral
ciprofloxacin 1000mg usa – buy septra for sale buy clavulanate sale
order lipitor 40mg pills atorvastatin 80mg for sale buy atorvastatin 10mg pills
写得真是太好了,我要保存上去当作条记,随时查看!