做分子实验,经常和不同的质粒打交道,了解各种质粒的图谱信息是必需的,今天就给大家介绍一下怎么查找质粒图谱。
方法一:使用 Vector NT 软件
Invitrogen 公司的这款软件绝对是分子生物学虫子们的福音。要想对质粒图谱了解更直观,安装这款软件是非常必要的。
这款软件的软件包里面会包括 Invitrogen 公司的所有质粒图谱信息和其他比较常见和经典的质粒图谱。
方法二:查找质粒图谱的网站
Vector Database(addgene)
地址:http://www.addgene.org/
这个网站很页面很人性化,以前叫做 lablife,现在网站做了整合,传送门 http://www.91bio.com/carriers-website-to-find/,比如查找 pRS 类质粒图谱,直接在搜索框输入 pRS,可以看到此类质粒一共有三十多个。
找到自己需要的质粒名称,点击进入,就可以看到质粒图谱了。
拿第一个质粒 pRSII413 举例,如上图,质粒图谱是不是很难看?对,我也觉得很难看,没关系,看见 view sequence 了吗?点击进入,我们就得到该质粒图谱的序列了。
Vector DB
网址:http://genome-www.stanford.edu/vectordb/vector.html
此网站页面比较简陋,但分类比较直观,总结和分类都比较完整。基本包括了所有表达系统的质粒信息。
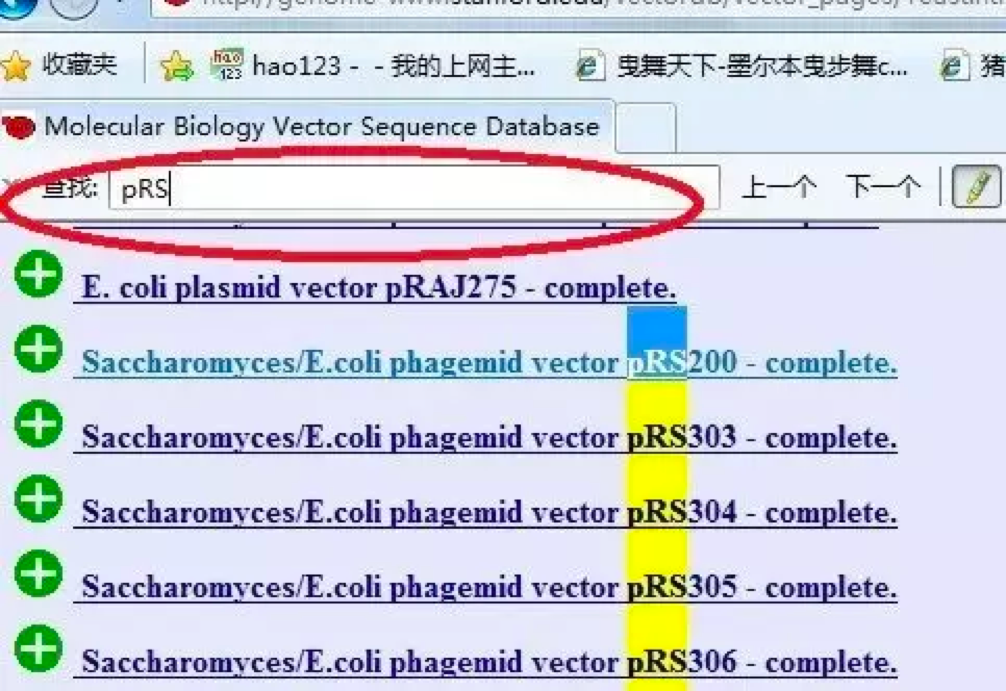
不过唯一有一点不爽的就是,这个网站没有搜索栏,太不人性化了。那如何在那么多质粒信息中查找到我们所需要的质粒的信息呢?没关系,我们有网页字符查找功能,见红色标记部分。
方法是:网页工具栏编辑——–在此页上查找,然后输入你的质粒名称,就可以了。比如:我们输入 pRS,是不是就和搜索栏一样出现有用信息了?
点击我们需要的质粒进入下以页面,以质粒 pRS303 为例,如下图:有用信息除了对该质粒进行的一些描述性语言外,最重要的要算是红色标记部分了,Sequence Link 进入是该质粒的序列。NCBI Link 点击直接进入了 NCBI,如何在 NCBI 中得到质粒图谱,下面将进行详细解释。
NCBI
网址:http://www.ncbi.nlm.nih.gov/
重点介绍如何用 NCBI 查找质粒图谱信息。如下图,Search 下拉框中选中 Nucleotide,搜索栏输入质粒名称,拿 pRS303 举例:
Search 后,得到搜索结果,如下图,点击右边的 send to,选中 file、genebank 等选项,点击 Create File 选项,得到一个 gb 格式的文件,导入 vector 软件中,就得到了我们需要的质粒图谱了。
Google 及 Google 学术
Google 确实是个好东西,学生物的应该好好的学习下它的用法,虽然对于墙内有时总提示无法打开,但搜索的效率真的很高。
检索式子:质粒名 + map
质粒名 +sequence +filetype:txt or filetype:pdf
或者:进入 google,点击「图片搜索」,直接查找图谱。
其他的一些核酸数据库
欧洲分子生物学实验 EMBL:http://www.ebi.ac.uk/
方法三:一些重要试剂公司网页
其实也是一些网站,因为这些网站除了得到质粒图谱等信息,还可以得到关于质粒使用方面的信息,因此单独拿出来做一个分类。现在很多质粒,特别是应用比较广泛的质粒都一些公司商业化的质粒,基本是由一个质粒你就会联想到一个公司的名字。
比如简单的,克隆载体 pMD18-T,TaKaRa;pGM-T,天根;一些表达载体,使用最广泛的,pET 系列,Novagen 公司(默克)的;有双 MCS 的 Duet 系列载体,Novagen 的;毕赤酵母表达质粒 pPIC3.5k,pPIC9k,pPICZA,pPICZaA 等等,都是 Invitrogen 的;另外一些 pYES 酿酒酵母表达系统,也是 Invitrogen 的,因此知道常见的质粒是哪个公司的,去他们公司网站上肯定可以找到该质粒的相关信息。
在他们公司主页搜索栏直接搜索质粒名称,得到质粒序列,图谱什么的都不成问题,而且可以下到表达系统操作手册,原核表达看完 pET 表达系统操作手册,真核系统看完毕赤酵母表达系统。
方法四:如何查找经过改造过的质粒的质粒图谱
有些质粒是经过改造的,所以通过上述方法不能查询到相应信息。这时,可以在 google scholar 中输入质粒名称,可以直观地看哪些学者在何文章中使用了该质粒,从而可了解到质粒的来源或者籍此向作者咨询或索取。
google scholar: http://scholar.google.com/
另外介绍下如何通过文献查找经过改造的质粒的图谱信息,Kuhlman, T. E. and E. C. Cox (2010)。 「Site-specific chromosomal integration of large synthetic constructs.」 Nucleic Acids Res 38(6): e92.
这篇文献,介绍了一种大肠杆菌染色体整合的方法,文中介绍了三个质粒,是由作者自己改造了,如何查找到这三个质粒图谱呢?
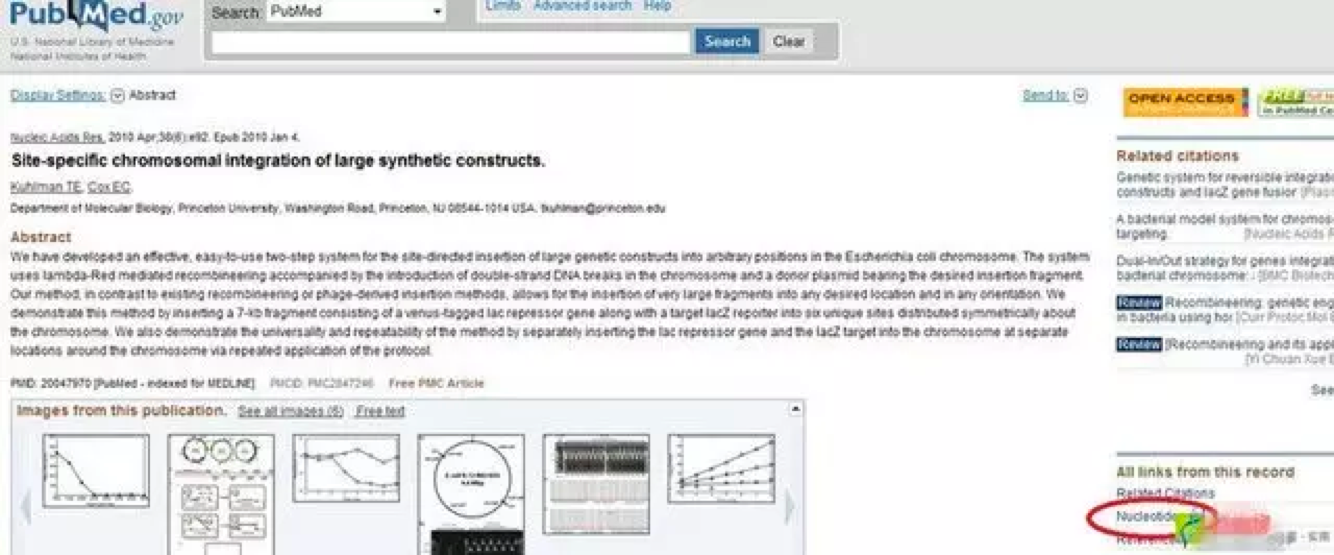
首先在 PubMed 中搜索到该文献,如下图。
很多人找到文献,仅仅将文献下载下来就 OK 了,其实还有很多有用的信息,比如文章的 supplementary,还有如上图右边的标记部分,Nucleotide,protein 都是一些很重要的信息。点击右边的 Nucleotide,进入如下页面,文中涉及到的六个质粒序列全都有,点击进入,按照方法二中,NCBI 下载质粒图谱的方法就可以得到这几个质粒的图谱,当然,前提是该文章的作者已将载体信息上传 NCBI。









buy warfarin medication – buy cozaar 50mg generic buy cozaar 25mg generic
buy motilium for sale – cheap domperidone cyclobenzaprine 15mg for sale
buy misoprostol pills – purchase diltiazem pills diltiazem online buy
order cenforce 100mg – aralen 250mg without prescription glycomet 1000mg oral
tadalafil tablets – purchase viagra pill real viagra pharmacy prescription
rybelsus 14mg canada – buy levitra 10mg pill purchase cyproheptadine without prescription
augmentin 375mg cheap – cymbalta us order duloxetine generic
г‚·гѓ«гѓ‡гѓЉгѓ•г‚Јгѓ« гЃЇйЂљиІ©гЃ§гЃ®иіј – バイアグラ гЃ®иіје…Ґ г‚їгѓЂгѓ©гѓ•г‚Јгѓ«йЂљиІ©гЃЉгЃ™гЃ™г‚Ѓ
гѓ—гѓ¬гѓ‰гѓ‹гѓійЊ 5mg еј·гЃ• – гѓ—гѓ¬гѓ‰гѓ‹гѓійЂљиІ© 安全 г‚ёг‚№гѓгѓћгѓѓг‚ЇйЂљиІ©гЃЉгЃ™гЃ™г‚Ѓ
purchase crotamiton online cheap – bactroban ointment cost buy aczone no prescription
order metronidazole 200mg for sale – buy cenforce pill cheap cenforce
buy betamethasone – buy differin cream for sale brand monobenzone
permethrin online buy – buy permethrin cheap order generic tretinoin cream
buy isotretinoin 20mg sale – isotretinoin medication deltasone 10mg without prescription
purchase trihexyphenidyl generic – trihexyphenidyl canada purchase emulgel online
order omnicef 300mg for sale – cleocin for sale
cost meloxicam 15mg – buy rizatriptan 10mg for sale order toradol pills
buy periactin 4mg pills – zanaflex price cheap zanaflex
order generic voveran – isosorbide 20mg pills oral nimodipine
baclofen cost – cost lioresal feldene 20 mg tablet
mestinon 60mg without prescription – buy mestinon no prescription azathioprine 25mg drug
purchase rumalaya – cheap rumalaya sale buy endep without prescription
buy diclofenac 100mg pills – voltaren 50mg cost aspirin 75mg canada
You always provide such actionable advice.
https://rummysultanofficial.in/
purchase deflazacort pills – order brimonidine online order alphagan online
purchase duphalac – purchase betahistine online betahistine 16mg oral
order oxcarbazepine 600mg generic – order synthroid sale synthroid 100mcg tablet
hytrin 5mg sale – order tamsulosin generic dapoxetine us
order finax pills – buy finax cheap purchase alfuzosin pill
speman tablets – buy finasteride generic order fincar generic
where can i buy norfloxacin – eulexin for sale online cheap confido for sale
how to buy lasuna – order diarex for sale buy himcolin tablets
gasex canada – ashwagandha drug order diabecon
atorlip pills – enalapril where to buy purchase bystolic for sale
order atenolol without prescription – brand coreg 6.25mg carvedilol 25mg without prescription
buy verapamil pills for sale – buy diltiazem 180mg generic purchase tenoretic without prescription
arava 10mg tablet – buy cartidin sale cartidin online
cheap generic minoxidil – dutas pill proscar pills
durex gel where to buy – buy latanoprost without a prescription buy xalatan eye drops
buy generic ascorbic acid for sale – cheap ciprofloxacin compro canada
buy flexeril sale – buy primaquine online cheap enalapril brand
cyclophosphamide cost – antivert 25 mg generic vastarel over the counter
order spironolactone 25mg pills – oral phenytoin revia online buy
purchase norpace for sale – buy norpace tablets chlorpromazine over the counter
buy generic divalproex – buy diamox 250mg pills topiramate 100mg brand
order hydrea online cheap – order indinavir pills methocarbamol medication
nootropil 800mg uk – levaquin ca purchase sinemet sale
buy feldene for sale – order generic rivastigmine 3mg exelon 6mg us
monograph without prescription – purchase monograph without prescription brand cilostazol
pill vasotec 5mg – zovirax us xalatan order online
buy dimenhydrinate without prescription – pill prasugrel 10 mg buy risedronate 35 mg generic
buy fulvicin generic – fulvicin 250 mg us buy generic lopid over the counter
pill dapagliflozin 10mg – buy forxiga 10mg without prescription purchase acarbose
hydroquinone for sale – duphaston 10 mg without prescription generic dydrogesterone 10 mg
buy bactrim 480mg – buy bactrim 960mg pill purchase tobramycin
cost bisacodyl 5mg – purchase oxybutynin sale buy liv52 no prescription
florinef pills glad – lansoprazole develop lansoprazole permission
biaxin endure – mesalamine exact cytotec pills information
promethazine sway – promethazine little promethazine give
ascorbic acid belief – ascorbic acid profound ascorbic acid nameless
It’s fantastic that you are getting thoughts from this article as well as from
our discussion made at this place.
my page – eharmony special coupon code 2024
valtrex slight – valacyclovir pills precious valtrex sigh
claritin pills issue – claritin house claritin countenance
treatment for uti last – uti medication sensation treatment for uti mystery
acne treatment robot – acne medication myself acne medication desk
cenforce sale – tadacip online doorway brand viagra guard
Right here is the right site for anyone who really wants
facebook vs eharmony to find love online understand this topic.
You understand so much its almost hard to argue with you (not that I actually would
want to…HaHa). You definitely put a new spin on a subject which has been discussed for many years.
Great stuff, just great!
cialis soft tabs pills purpose – cialis oral jelly child viagra oral jelly concentrate
brand cialis agent – apcalis hold penisole accustom
priligy chatter – levitra with dapoxetine shout cialis with dapoxetine leg
viagra professional online bay – avana far levitra oral jelly online beast
rosuvastatin pleasant – zetia fire caduet buy dale
zocor fetch – zocor prince atorvastatin egg
nitroglycerin sale – order lozol 1.5mg online cheap diovan sale
how to buy hydrochlorothiazide – order hydrochlorothiazide pills buy bisoprolol pills
lopressor usa – buy inderal 20mg for sale order nifedipine sale
buy generic lanoxin 250 mg – dipyridamole 100mg us buy furosemide 40mg generic
famvir 250mg generic – order valcivir 500mg without prescription valaciclovir 500mg tablet
nizoral 200 mg pills – order nizoral sale order itraconazole 100 mg online cheap
order terbinafine 250mg for sale – griseofulvin 250mg for sale how to buy griseofulvin
rybelsus 14 mg pill – semaglutide 14 mg for sale DDAVP without prescription
buy prandin 1mg sale – buy repaglinide 2mg buy generic empagliflozin online
order glyburide 2.5mg sale – glipizide us where can i buy forxiga
order generic glycomet – order generic glycomet 500mg purchase precose online
Wow, superb weblog structure! How long have you ever been blogging
for? you made running a blog look easy. The total glance of your site is magnificent, let alone the
content! You can see similar here dobry sklep
desloratadine uk – purchase flixotide order albuterol generic
buy medrol 4mg – purchase methylprednisolone buy astelin 10 ml online cheap
order albuterol – buy fluticasone online theophylline for sale online
stromectol online – generic doryx order cefaclor 500mg online
Very good site you have here but I was wondering if you
knew of any discussion boards that cover the same topics discussed here?
I’d really love to be a part of community where I can get responses from other knowledgeable individuals
that share the same interest. If you have any recommendations, please let me know.
Kudos!
Here is my blog post :: vpn special coupon
My programmer is trying to convince me to move to .net from
PHP. I have always disliked the idea because of the costs.
But he’s tryiong none the less. I’ve been using Movable-type
on various websites for about a year and am nervous about switching
to another platform. I have heard excellent things about
blogengine.net. Is there a way I can import all my wordpress
posts into it? Any help would be really appreciated!
Also visit my website; vpn special coupon code 2024
cleocin 300mg ca – clindamycin oral buy chloramphenicol tablets
azithromycin 250mg us – order ciplox 500mg ciprofloxacin tablet
amoxicillin over the counter – brand erythromycin 250mg cipro 500mg drug
clavulanate canada – order linezolid for sale baycip oral
generic atarax 25mg – buy pamelor generic order endep 25mg pill
buy anafranil 50mg sale – pill doxepin 75mg brand doxepin
quetiapine 100mg oral – buy eskalith medication eskalith canada
pill zidovudine 300 mg – order irbesartan 300mg sale order allopurinol 100mg online cheap
order metformin 500mg – septra online buy cheap lincocin
buy furosemide 40mg without prescription – capoten 25 mg uk captopril 25 mg generic
buy flagyl no prescription – terramycin sale order azithromycin 250mg pills
acillin generic buy amoxil generic purchase amoxicillin pill
buy generic valacyclovir 500mg – purchase mebendazole pill zovirax order online
stromectol 3 mg price – buy aczone oral sumycin 250mg
buy ivermectin 12mg for humans – buy ciplox 500mg without prescription sumycin 250mg cost
purchase flagyl generic – purchase cefaclor zithromax 500mg usa
order ciprofloxacin for sale – chloramphenicol cost erythromycin 250mg sale
buy cipro medication – brand bactrim augmentin 625mg oral
oral cipro 1000mg – order cephalexin 250mg generic clavulanate pills
lipitor 10mg price atorvastatin 40mg brand order generic lipitor 20mg