6. 文件读取
最常用的就是read.table(),读进去就是data.frame。
让我们来看看read.table()有哪些参数吧。命令行输入:
??read.table()
输入后回车,跳转到help界面。可以看到如下用法信息。
read.table(file, header = FALSE, sep = "", quote = "\"'", dec = ".", numerals = c("allow.loss", "warn.loss", "no.loss"), row.names, col.names, as.is = !stringsAsFactors, na.strings = "NA", colClasses = NA, nrows = -1, skip = 0, check.names = TRUE, fill = !blank.lines.skip, strip.white = FALSE, blank.lines.skip = TRUE, comment.char = "#", allowEscapes = FALSE, flush = FALSE, stringsAsFactors = default.stringsAsFactors(), fileEncoding = "", encoding = "unknown", text, skipNul = FALSE)
具体看看一些比较重要的参数
header=FALSE : 改为TURE后,将第一行的值作为列名。如果第一行就是数据的话,可以先读取后用colnames()加上。
sep=””:指定分隔符,默认无,一般遇到的类型,tab/tsv文件”\t”,csv文件用的是逗号分隔符”,”,还可能有空格分隔符。分隔符指定不正确的话,可能会使得数据读取错误。
quote=”\”‘”:quote制定包围字符型数据的字符。字符一般是由双引号或单引号括起的。这在存写文件时要注意使quote=””,之前遇到把基因组坐标当成字符串输出的情况,使得其他软件无法识别。
dec = “.”: 指定小数点
row.names: 可指定某一列作为行名,e.g.: 指定第一列为行名,row.names = 1
as.is = !stringsAsFactors :as.is 字符向量是否转换成因子
nrows = -1 最大读入行数,即读入前多少行,有时候遇到数据量过大,需要只读取部分时使用。“-1”表示都读入
skip = 0 :skip = n时,跳过文件的前n行fill = !blank.lines.skip 从一个电子表格中导出的文件通常会把拖尾的空字段忽略掉。为了读取这样的文件,必须设置参数 fill = TRUE
此外还有,read.csv(),读取csv文件,等等。
练习用的文件内容如下,复制粘贴到txt中保存为exp.txt。
Gene,rp1,rp2,rp3
gene1,5,6,5
gene2,20,30,25
gene3,100,90,105
gene4,8,7,8
gene5,13,14,15
gene6,24,20,30
gene7,70,80,70
gene8,6,7,9
gene9,3,4,2
gene10,0,0,0
练习一下吧,看看两者的区别。
setwd("E:/CODE/R") data1 <- read.table(file="exp.txt", header = TRUE, sep = ",", row.names = 1, fill = TRUE) data1
## rp1 rp2 rp3 ## gene1 5 6 5 ## gene2 20 30 25 ## gene3 100 90 105 ## gene4 8 7 8 ## gene5 13 14 15 ## gene6 24 20 30 ## gene7 70 80 70 ## gene8 6 7 9 ## gene9 3 4 2 ## gene10 0 0 0
data2 <- read.table(file="exp.txt", header = TRUE, sep = ",", fill = TRUE) data2
## Gene rp1 rp2 rp3 ## 1 gene1 5 6 5 ## 2 gene2 20 30 25 ## 3 gene3 100 90 105 ## 4 gene4 8 7 8 ## 5 gene5 13 14 15 ## 6 gene6 24 20 30 ## 7 gene7 70 80 70 ## 8 gene8 6 7 9 ## 9 gene9 3 4 2 ## 10 gene10 0 0 0
7. Apply家族的成员们
apply家族有很多成员,第七个问题来介绍他们吧。
首先是老大,apply()。
apply(X, MARGIN, FUN, ...) # X, 数组(array),包含矩阵(matrix) # MARGIN, 可填 1,2,c(1,2)。1代表行,2代表列,c(1,2)同时对行和列都进行操作。 # FUN,函数,即要进行的操作。可以是常见的sum,mean,也可以是自己写的自定义函数。
举个栗子:换个方法实现第三问提到的rowMeans, rowSums。
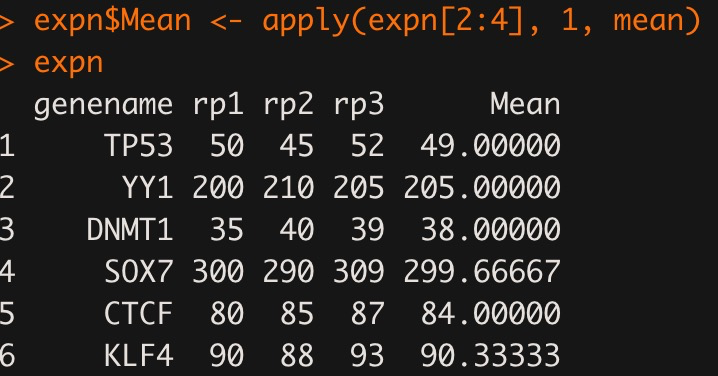
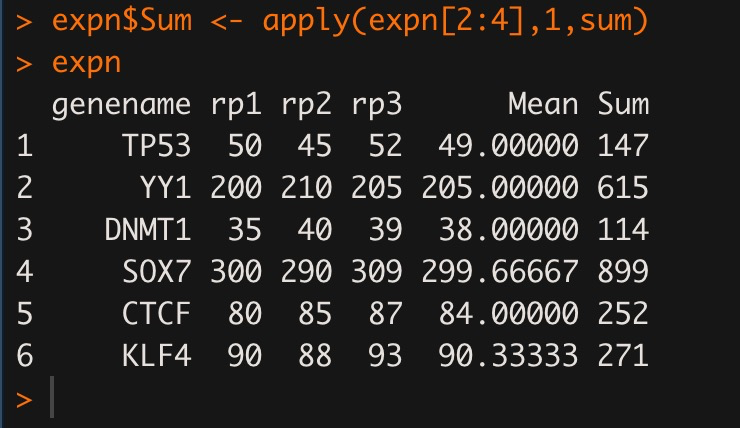
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),rp1=c(50,200,35,300,80,90),rp2=c(45,210,40,290,85,88),rp3=c(52,205,39,309,87,93)) expn #温馨提示,这里是因为数据不多的情况下才输入变量名,数据量很大的情况下,还是head(expn)哦 expn$Mean <- apply(expn[2:4], 1, mean) expn expn$Sum <- apply(expn[2:4],1,sum) expn
看看结果
lapply():
l是list的意思,处理的对象可以是list,也可以是data.frame。
一般lapply对list的使用频率较低,对data.frame使用频率较高,会自动把数据框按列进行分组,再进行计算,并返回一个list。
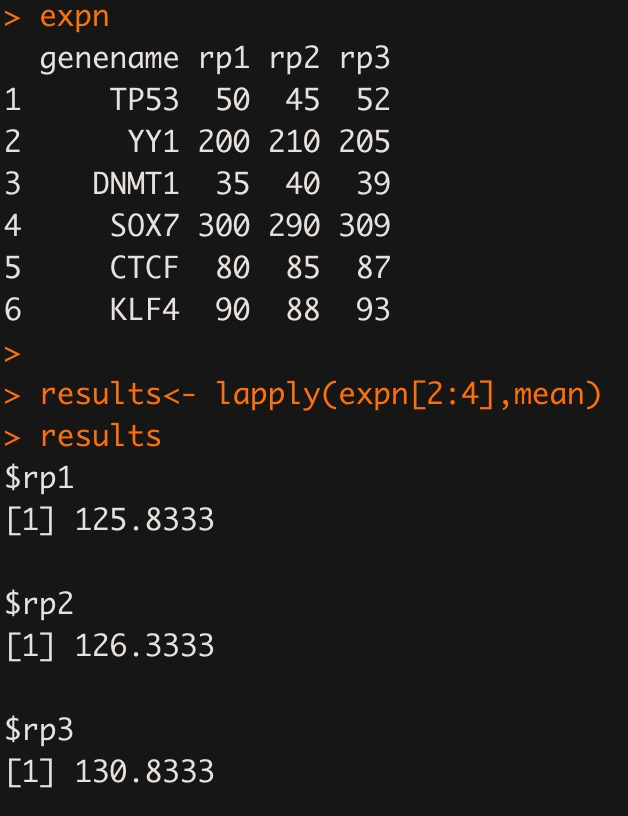
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),rp1=c(50,200,35,300,80,90),rp2=c(45,210,40,290,85,88),rp3=c(52,205,39,309,87,93)) expn results<- lapply(expn[2:4],mean) results
可以看到,results是对每个rp进行对处理。这里与colMeans, colSums的效果是一致的。
sapply():
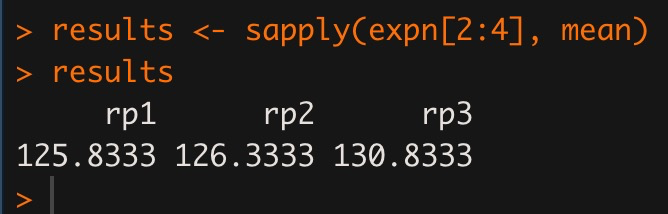
sapply与lapply用法类似,但是返回为vector。
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),rp1=c(50,200,35,300,80,90),rp2=c(45,210,40,290,85,88),rp3=c(52,205,39,309,87,93)) expn results <- sapply(expn[2:4], mean) results
tapply():
tapply也同样是对数据进行批量操作,但是是根据分组情况进行的计算。因此我们在使用这个函数时,需要指定分组信息,而且必须是factor格式。
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4","TP53", "YY1","DNMT1","SOX7","CTCF","KLF4","TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"), exp=c(50,200,35,300,80,90,45,210,40,290,85,88,52,205,39,309,87,93), group=c("rp1","rp1","rp1","rp1","rp1","rp1","rp2","rp2","rp2","rp2","rp2","rp2","rp3","rp3","rp3","rp3","rp3","rp3")) expn mean.results <- tapply(expn$exp,expn$genename, mean) mean.results sum.results <- tapply(expn$exp,expn$genename, sum) sum.results
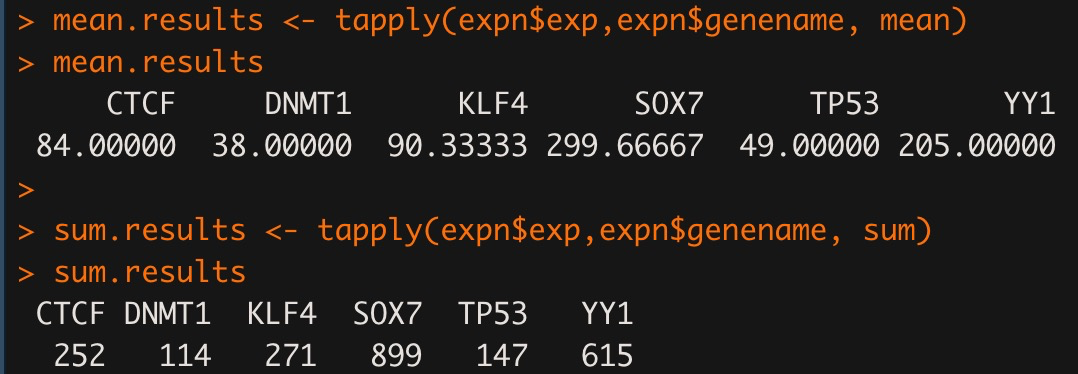
此外,apply家族函数还包含mapply和vapply等,就不展开那么多了。
8. GO/KEGG 富集分析
假设你有一串genelist。
gene 1
gene 2
gene 3
…
GO/KEGG富集的实质是什么呢?就是我们想看这个基因集中的基因在各个GO/KEGG基因集的分布是否存在一定的偏好性,即这个基因集中的基因是否集中在某几个GO/KEGG中,这样的分布是否显著性,而非随机性。这就需要统计学检验分析,这里运用到的Fisher精确检验。
无论是做什么分析都好,最关键的是要弄清楚背后的原理,目的,还有统计学分析手段区别。代码只是帮助我们把分析流程化。当然,比起网页工具,代码的好处在于可批量重复操作,可DIY性高。
R中最热门的做GO/KEGG分析的强大工具,就是clusterProfiler了。
话不多说,看代码。
# GO分析 setwd("/Users/meijun/Documents") library("clusterProfiler") library("org.Mm.eg.db") genelist <- read.table(file="/Users/meijun/Documents/genelist.txt", sep = "\t", header = F,fill = TRUE) head(genelist)#查看数据类型,读进去是data.frame ego <- enrichGO(gene = genelist$V1, OrgDb = org.Mm.eg.db, ont = "ALL", pAdjustMethod = "BH", pvalueCutoff = 0.01, qvalueCutoff = 0.05, keyType = "SYMBOL") dotplot(ego, title = "GO enrichment analysis") #画图 write.csv(ego, file = "GO.csv") #保存文件 #enrichGO() #OrgDb需要根据物种进行选择 #ont是可以选择的,CC(cell component),BP(biological process), MF(molecular function),ALL (包含前面三个) #pAdjustMethod,多重检验校正的手段,默认是最严格的BH(Benjamini & Hochberg,人名) #pvalue,即Fisher精确检验计算的p值,这里设定的是阈值 #qvalue,多重检验校正后的pvalue,区别于pvalue。 #keyType, 输入的gene id类型。
练习文件(自行下载保存):?genelist.txt
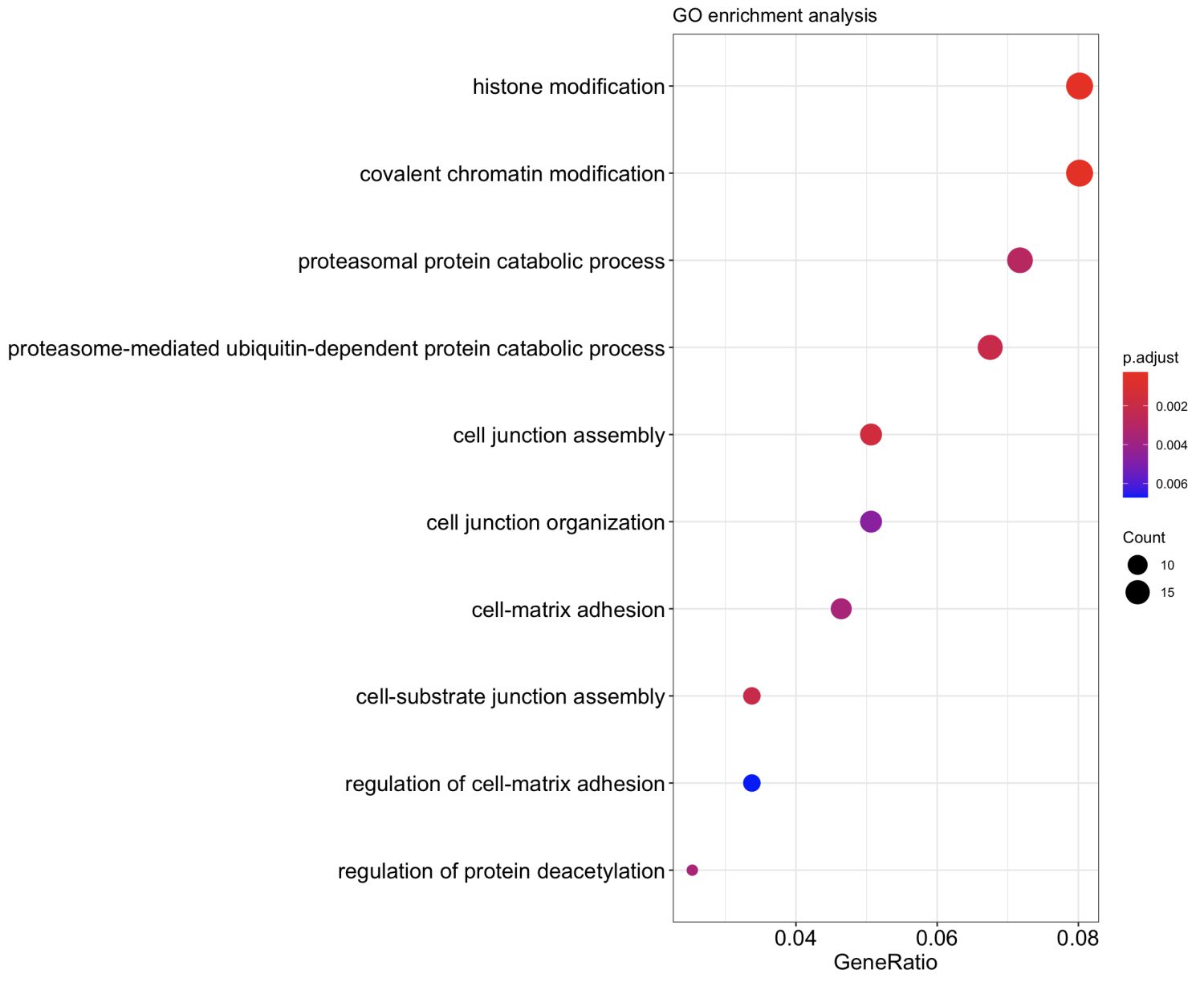
作业:研究clusterProfiler作者写的manual(https://yulab-smu.github.io/clusterProfiler-book/),然后完成KEGG的分析吧。提醒一下,KEGG要求输入的gene id类型必须是Entrez ID,我提供的是SYBMBOL,这样的话,需要做个ID conversion哦。
9. 求反向互补序列
revSequence <- function(seq_list){ paste(rev(unlist(strsplit(seq_list,NULL))),collapse="") } comSequence <- function(seq_list){ seq_list <- gsub("A", "N", seq_list) seq_list <- gsub("T", "A", seq_list) seq_list <- gsub("N", "T", seq_list) seq_list <- gsub("C", "H", seq_list) seq_list <- gsub("G", "C", seq_list) seq_list <- gsub("H", "G", seq_list) seq_list } rev_comSequence <- function(seq_list){ seq_list <- gsub("A", "N", seq_list) seq_list <- gsub("T", "A", seq_list) seq_list <- gsub("N", "T", seq_list) seq_list <- gsub("C", "H", seq_list) seq_list <- gsub("G", "C", seq_list) seq_list <- gsub("H", "G", seq_list) paste(rev(unlist(strsplit(seq_list,NULL))),collapse="") } indexlist <- data.matrix(c("ATCC","CTGA")) indexlist revcomindexlist <- as.matrix(apply(indexlist, 1, rev_comSequence)) revcomindexlist
遇到index要求反补的问题,看到有用python写的,我偏向用R,于是用R写了几个function。反序还好,写互补的时候,只能用简单粗暴的交叉替换了(丢人,但是能用)。
10. 画个boxplot
请移步:https://www.yuque.com/docs/share/6e0d9e89-2baa-4fa6-acd6-2ee57a1fa866?#
内容是很早之前的了。



