1. Bio3D的介绍
Bio3D 是一款依托于 R 语言的工具包,包含用于分析生物分子结构、序列和轨迹数据的实用程序。功能包括读取和写入生物分子结构、序列和动态轨迹数据的能力,执行原子选择、重新定向、叠加、刚性核识别、聚类、距离矩阵分析、守恒分析、正常模式分析和主成分分析。
2. Bio3D的安装
本机Window10系统
Bio3D下载链接来源:
http://thegrantlab.org/bio3d/articles/online/install_vignette/Bio3D_install.html
先不急着安装,需要先下载R语言:
https://bitbucket.org/Grantlab/bio3d/downloads/
安装并打开R语言,输入:
install.packages(“bio3d”, dependencies=TRUE)【详细解释见:https://rdrr.io/r/utils/install.packages.html】
出现界面如下:
选择一个工作镜像(China内)即可完成安装
检测是否成功安装:输入help(package=”bio3d”)出现“关于编辑包‘bio3D’ 的信息”即为成功安装。
3. Bio3D应用实例
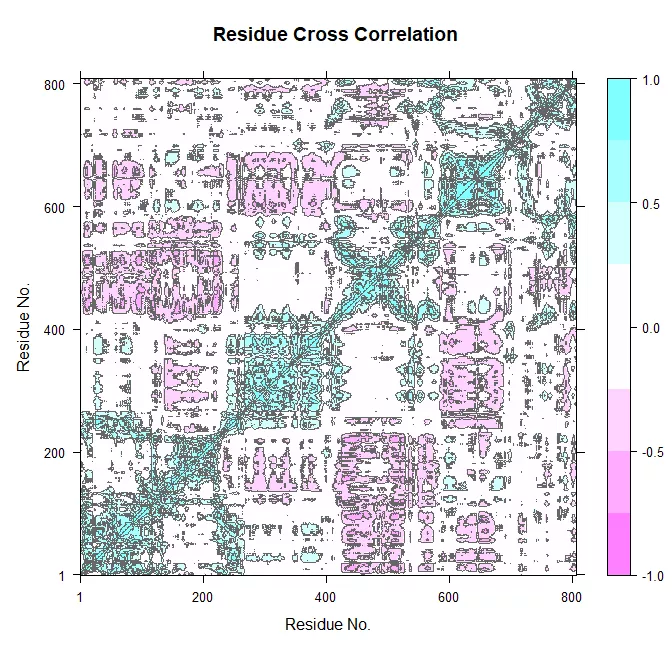
以Bio3D计算DCCM作为应用实例。
动态相关性矩阵(Dynamic cross-correlation matrices, DCCM),表示蛋白质中每个氨基酸的特定原子,比如Cα原子和其它氨基酸的Cα原子之间的相关性,提供蛋白质在大尺度范围内相关运动的一些信息。DCCM计算数值的取值范围从完全负相关的-1.0到完全正相关的+1.0。越接近数值1表示相关性越强,正负表示两个原子运动方向相同(反)。
首先需要准备两个文件.dcd和.pdb(以taq DNA聚合酶为例)
打开R语言
library(bio3d)
require(bio3d); require(graphics);
pause <- function()
cat(“Press ENTER/RETURN/NEWLINE to continue.”)
pause()
trtfile <- system.file
(“D:/Program Files/R/R-4.1.0/bin/x64/start.dcd”,package=”bio3d”)
dcd <- read.dcd(“D:/Program Files/R/R-4.1.0/bin/x64/start.dcd”) (#定位到.dcd和.pdb所在的文件夹)
pdbfile <- system.file
(“D:/ProgramFiles/R/R-4.1.0/bin/x64/start.pdb”,package=”bio3d”)
pdb <- read.pdb(“D:/ProgramFiles/R/R-4.1.0/bin/x64/start.pdb “)
print(pdb)
pause()
dim(dcd)
ncol(dcd) == length(pdb$xyz)
pause ()
ca.inds <- atom.select(pdb, elety = “CA”)
xyz <- fit.xyz(fixed = pdb$xyz, mobile = dcd,
fixed.inds = ca.inds$xyz,
mobile.inds = ca.inds$xyz)
DCCM的计算:
cij <- dccm(xyz[,ca.inds$xyz])
plot(cij)
RMSD的计算(表示整个结构随时间的位置差异):
rd <- rmsd(xyz[1,ca.inds$xyz], xyz[, ca.inds$xyz])
plot(rd, typ= “l”, ylab = “RMSD”, xlab = “Frame No.”)
points(lowess(rd), typ =”l”, col = “red”, lty = 2, lwd = 2)
summary(rd)
pause()
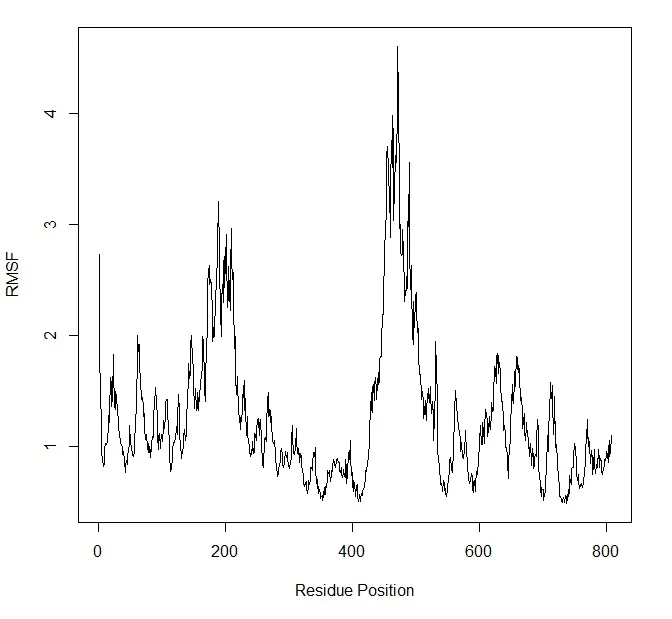
RMSF的计算 (表示单个氨基酸残基的柔度,或计算模拟过程中特定残基移动的程度):
rf <-rmsf(xyz[, ca.inds$xyz])
plot(rf, ylab =”RMSF”, xlab = “Residue Position”, typ=”l”)
pause()
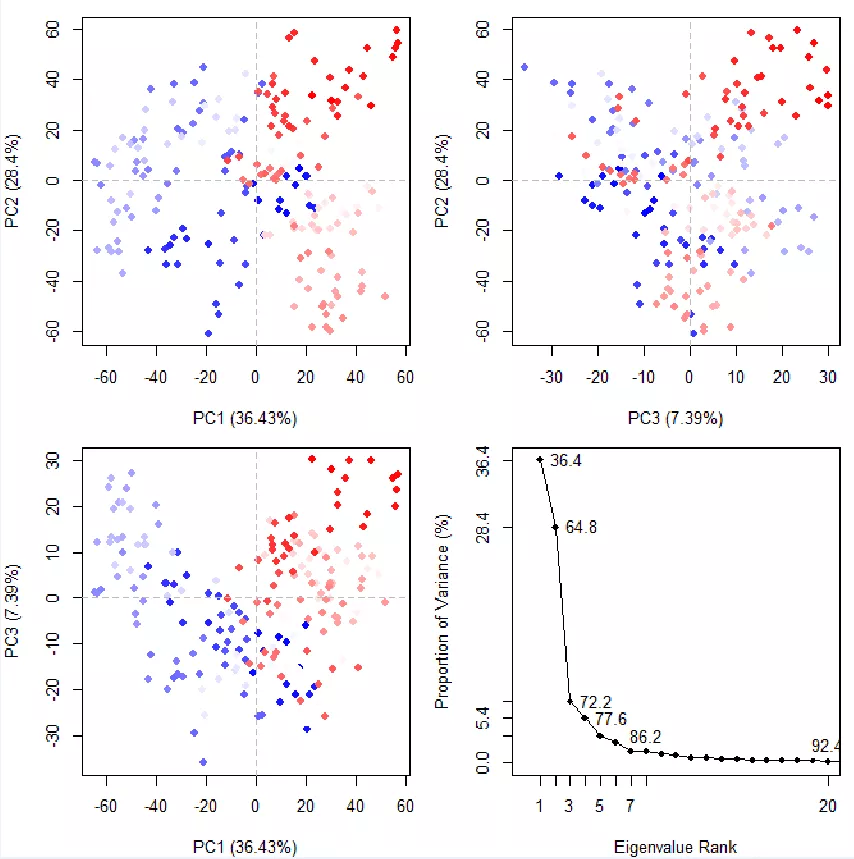
PCA的计算(Principal ComponentAnalysis,主成分分析)
pc <- pca.xyz(xyz[,ca.inds$xyz])
plot(pc, col= bwr.colors(nrow(xyz)))
备注:.dcd文件可以通过VMD来获得。即
先将GROMACS得到的last10ns.xtc转为start.pdb
gmxtrjconv -s md.tpr -f last10ns.xtc -o start.pdb -dump 0 (选择1, protein)
打开VMD,将start.pdb转存为start.dcd












order digoxin 250mg online – lanoxin 250mg drug purchase lasix sale
famciclovir 500mg pill – buy generic valcivir 500mg valcivir 500mg usa
buy ketoconazole pills – lotrisone generic sporanox usa
buy semaglutide 14 mg generic – glucovance brand order DDAVP for sale
terbinafine without prescription – buy diflucan paypal griseofulvin usa
glycomet 1000mg canada – acarbose medication buy precose pills
repaglinide canada – repaglinide 2mg ca jardiance price
Wow, fantastic blog format! How lengthy have you ever been running a blog for?
you make running a blog look easy. The full glance of your
site is great, as neatly as the content! You can see similar here sklep online
buy clarinex 5mg sale – albuterol over the counter purchase ventolin inhalator generic
stromectol cost – cefaclor online buy cefaclor 250mg without prescription
ventolin 2mg oral – buy albuterol 2mg online order theophylline 400 mg generic
yaEDsJcmF
buy azithromycin 500mg for sale – ofloxacin for sale online brand ciplox 500 mg
Hi i am kavin, its my first time to commenting anyplace,
when i read this piece of writing i thought i could also make comment due to this sensible paragraph.
Look into my web page vpn coupon code 2024
cleocin price – terramycin price buy chloromycetin pills for sale
buy cheap generic augmentin – purchase septra online ciprofloxacin 1000mg ca
amoxicillin canada – order keflex 500mg sale baycip over the counter
buy atarax 25mg without prescription – hydroxyzine 25mg ca buy endep pill
cheap clomipramine 50mg – buy sinequan 75mg generic where to buy doxepin without a prescription
order seroquel 50mg pill – buy cheap generic ziprasidone buy eskalith pill
clozaril 50mg usa – cheap famotidine 20mg famotidine pills
purchase zidovudine sale – order glucophage online cheap buy allopurinol pills for sale
buy metformin 500mg generic – glycomet medication lincocin without prescription
furosemide tablet – capoten order buy capoten tablets
oral ampicillin monodox brand buy amoxicillin generic
metronidazole online buy – azithromycin 250mg canada buy azithromycin 250mg online cheap
stromectol usa – cefuroxime order sumycin 250mg sale
how to get valtrex without a prescription – buy starlix pills for sale zovirax generic
purchase ciprofloxacin online – erythromycin buy online order erythromycin generic
where to buy flagyl without a prescription – order generic oxytetracycline purchase zithromax generic
buy ciprofloxacin 500mg sale – buy cipro 500mg online order augmentin 1000mg online cheap
cipro 1000mg ca – buy augmentin generic order generic augmentin 1000mg
order atorvastatin 20mg for sale lipitor 20mg canada cheap lipitor 20mg
大神,请问得到动态相关性矩阵的csv文件后,怎么用originpro画这种热图呀
请问您解决这个问题了吗?我想请教一下