基本原理
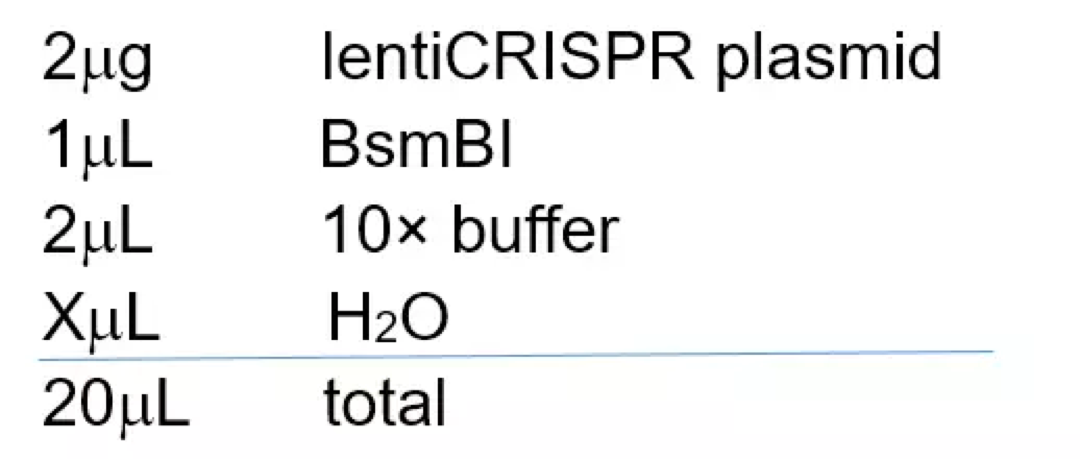
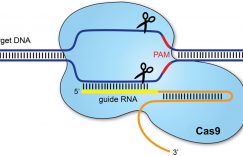
CRISPR-Cas9是一种细菌获得性免疫,用于降解入侵的外源DNA。CRISPR RNA (crRNA) 与转录激活crRNA (Trans-activating crRNA, tracrRNA) 退火形成的复合物能特异性识别与其互补的基因组序列,引导Cas9核酸内切酶在目的片段生成DNA双链断裂,如下图所示:

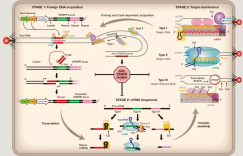
通过基因工程手段对crRNA和tracrRNA进行改造,将其连接在一起得到sgRNA (single guide RNA)。通过将表达sgRNA的原件与表达Cas9的原件相连接,得到可以同时表达两者的质粒,将其转染细胞,便能够对目的基因进行基因操作,在PAM (5’-NGG) 上游 ~3bp进行切割,如下图所示:
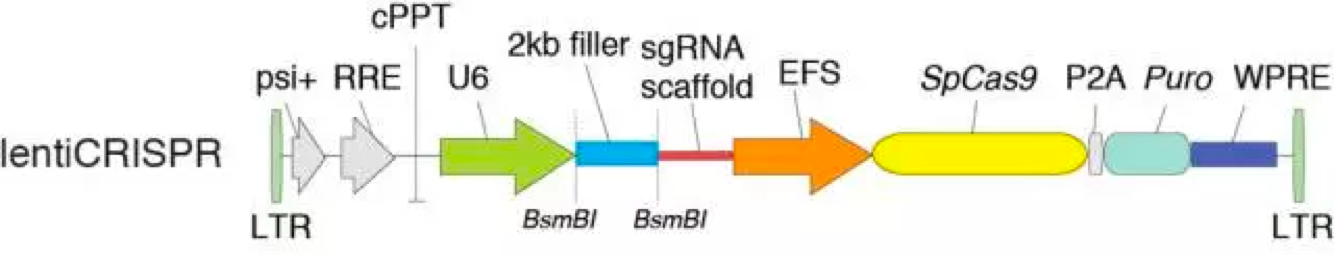 因此本实验的关键步骤是设计一对20bp(不包括NGG在内的)完全互补的oligo插入到如上图所示的filler。另U6启动子需要5’端的G起始转录,因此若设计的oligo第一个碱基非G,需额外增加一个G。
因此本实验的关键步骤是设计一对20bp(不包括NGG在内的)完全互补的oligo插入到如上图所示的filler。另U6启动子需要5’端的G起始转录,因此若设计的oligo第一个碱基非G,需额外增加一个G。
详细操作流程
1. 设计sgRNA
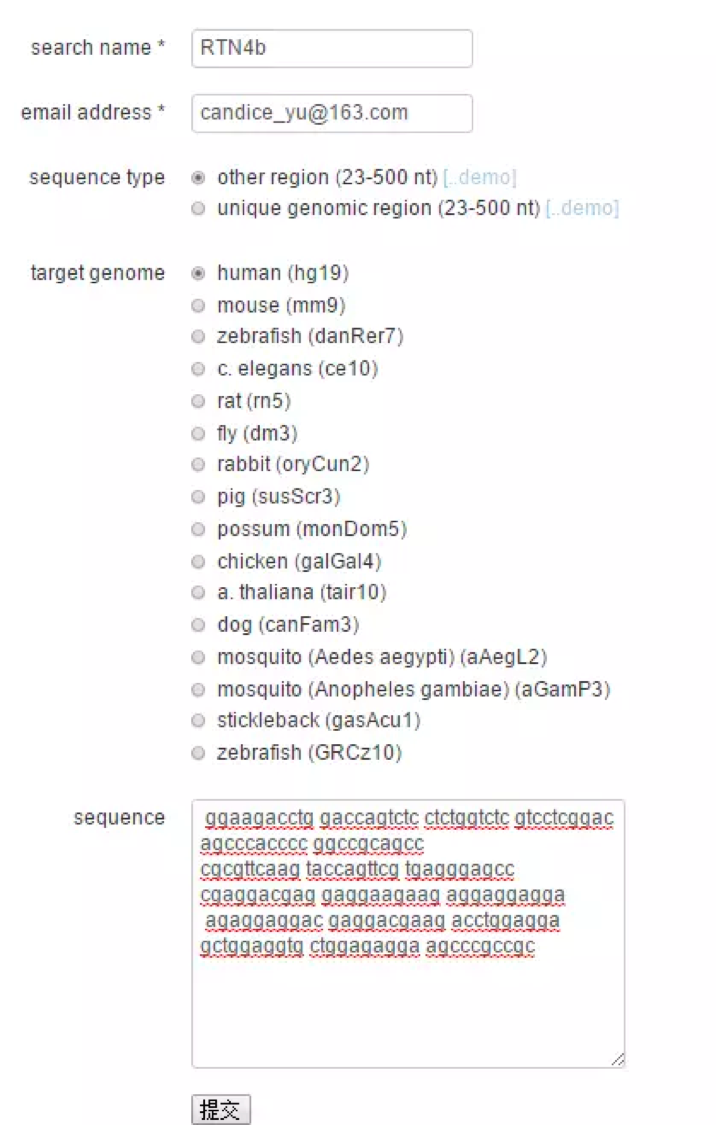
确定靶基因的序列后,可通过在线网站设计(http://tools.genome-engineering.org ),或根据靶基因的CDS序列自行设计,最方便的是在human KO Library sgRNA里选择。无论选择哪种方式,都建议进行一下blast。
附上在线设计网页截图。输入基因的name,填写email address,设计结果稍后会发送到此邮箱,选择序列的类型以及物种。如sequence type选择unique region,一次只能输入23~500bp的基因片段,最好一次只输入一个外显子,避免guide序列跨越内含子,都选择键入后点击提交,关注邮箱或者在线等。
分析中,显示你所键入的基因序列里有36对可选的guide序列。
分析结束,点击Download as genbank查看结果,此时也会收到邮件。
选择分数较高的Guide序列,以Guide#1序列为例,2条单链oligo的序列如下:oligo1:5’- caccGCGTTCAAGTACCAGTTCGTG -3’; oligo2:5’- aaacCACGAACTGGTACTTGAACGc。标粗部分与经BsmBI酶切后的载体互补配对。BsmBI又名BbsI。
※oligoDNA序列的第一个碱基必须是G,如选取的Guide序列的第一个碱基不是G,需自行添加一个额外的G。
2. Oligo退火形成duplex
PCR仪 95℃ 5 min,缓慢降温至室温1h,1:200稀释duplex。
3. 质粒酶切
4. 胶回收
大片段约11kb 回收;小片段约2kb为切下来的filler,不要。
5. 连接
室温连接4-6 h。
6. 转化(Amp+),挑克隆,摇菌,抽提质粒,测序!
7. 转染
24孔板,细胞不要过满,同实验室日常转染操作,可选择性设置三个平行。同时转染空载作为阴性对照。每孔500 ng。
8. puromycin筛选
转染稳定48-72h后加puromycin进行筛选,1~3g/mL,直至不再有细胞数量稳定,不再有细胞因不耐药死去。
9. 单细胞分离
由于篇幅原因,有限稀释法具体细节会在后面的文章单独讲解,请关注科研小助手后续文章。100个细胞铺在一个大盘或96孔板中。剩余的细胞冻存。
10. 获得单克隆细胞株
当肉眼可见类似于菌落的细胞团时,将其从大盘或96孔板消化下来转移至12或24孔板中继续扩大培养。注意不要污染到其它细胞团,不要选取过密的细胞团。
11. 扩大培养,提取细胞全基因组DNA和细胞总蛋白
12. 测序
Guide序列上游100bp左右和下游200bp左右为上下游设计一对引物,以全基因组为模板,以上述引物进行PCR。PCR产物送测序,或将产物用T7E1进行酶切消化检测突变。
13. Western
与阴性对照相比,靶基因应完全敲除








It’s impressive that you are getting thoughts from this article as well as from our dialogue made at this time.
You have made some decent points there. I checked on the web for additional information about the issue and found most people will go along with your views on this web site.
Do you have a spam problem on this website? I also am a blogger, and I was wondering your situation; we have developed some nice practices and we are looking to exchange techniques with other folks, be sure to shoot me an email if interested.
It is really a nice and helpful piece of info. I’m happy that you simply shared this useful info with us. Please keep us up to date like this. Thanks for sharing.
bppumps.co.in
Hello, Neat post. There is an issue with your website in web explorer, maytest this? IE nonetheless is the market chief and a large element of other people will leave out your magnificent writing due to this problem.
[url=https://brasseri1864.se/offre-favorable-avec-1xbet-mobile/]brasseri1864.se[/url]
Superb info. Appreciate it.
gta 5 online casino update captain cooks casino canada casinos online 2022
u1bssk
With thanks, Awesome stuff.
best online casino names free online casino slot machines fotos de casino online
With thanks. Wonderful information.
beste online casino ohne 5 sekunden regel real money online casino canada online casino games rigged
Cheers, A lot of write ups.
online casino mit 10 euro mindesteinzahlung online casino deposit bonus beat online casino games
aja6l2
Appreciate it. Quite a lot of facts!
online casino games for real money in india biggest online casino online casinos ohne download
Incredible a lot of beneficial data.
online casinos mit schneller auszahlung Г¶sterreich win real money online casino online casino worldwide
Thanks, Plenty of info!
new online usa casinos 2022 online casinos canada new jersey online casino promotions
You mentioned this wonderfully.
online casino handy best casino slot online dansk online casino
Nicely put. Thanks.
mega 7 online casino spin casino canada wildz casino online
Amazing a good deal of wonderful knowledge.
online casino in india is legal online free slots casino best online casino australia real money
You said it fantastically.
online casino gambling legal online casino games real money no deposit brst online casino
Terrific content. Thank you.
gta 5 online diamond casino free casino online slots free spins sign up online casino
You actually stated that very well.
hollywood online casino pa casino online slots best casino online in canada grand mondial
Kudos, Quite a lot of material.
online casinos that accept discover online casinos canada bet mgm online casino phone number
Great info. Thanks a lot.
any new online casinos casino rewards canada fairgo online casino australia
Thanks a lot. Plenty of posts.
australia casino online slot machine casino online online casino bonus deutschland
Very well voiced without a doubt. .
online casino 1x turnover online free casino slots bentley 88 online casino
Fine posts. Appreciate it!
nc online casino real money casino online fanduel michigan online casino
Appreciate it. Plenty of content!
best payout online casino real money usa real money casino online list all new usa online casinos
You actually revealed this very well.
online casino blackjack real money casino online games free asian sic bo online casino
Amazing content, Appreciate it!
cash online casino best online real money casino online casino no verification withdrawal uk
Thanks a lot! Great information!
australian online waterwheel casinos las vegas online casino free slots casino online colorado
You’ve made your point.
ruleta online casino gran madrid casinos in ontario canada online blackjack live casino
Truly all kinds of very good material!
t-rex online casino real casino slots online citi casino online
Truly quite a lot of amazing advice.
best online casino fast withdrawal online casino win real money online casino magyarorszag
“I thought you usually showered after practice. In the locker room.” His dad asks as he walks into the open door of the bathroom.
You made the point.
online casino slots nj best online casino slot machines gta online casino heist change vault contents
You’ve made your position pretty well..
888slots online casino online live casino best online canadian casino
Many thanks! An abundance of data.
online casino slot machine slots casino online which online casino is the best
“Pick it up and smell the crotch.” His dad says. “What are you now, son?”
You mentioned this very well.
online casino with best payout [url=https://usagamblinghub.com/#]new online casinos usa real money[/url] best blackjack online casino
Wow quite a lot of fantastic facts.
real money online casinos michigan [url=https://usacasinomaster.com/#]no deposit online casino[/url] afghanistan online casinos
Kudos. Valuable stuff.
online casino real money australia [url=https://luckyusaplay.com/#]brand new online casinos usa[/url] ocean ac online casino
Awesome information. Many thanks!
ocean online casino promo code [url=https://luckyusaplay.com/#]online gambling casino[/url] new online casino west virginia
“What…huh…what, dad?” He asks, puzzled by the question from his dad. As his cock is mere inches from his dad’s face. [url=https://arturzasada.pl/]gej porno[/url] Garrett does not go hog-wild on his tool. He caresses and adores it like one would an idol.
You stated this effectively!
agen judi igkbet casino online legit online casinos 2022 no deposit bonus online casino
This is nicely said. .
online casino in cambodia casinos online online casinos in alberta
You revealed this really well.
best georgian online casino site online casino sign up bonus cats online casino game
Perfectly expressed indeed. !
casino sports betting online [url=https://uscasinoguides.com/#]casino online casino[/url] amazon queen online casinos
Truly a good deal of excellent advice!
polskie casino online [url=https://usaplayerscasino.com/#]casino games free online[/url] efbet casino online igri
Thanks! Plenty of content.
casino de barcelona online online casino free play online casino deutschland verboten
With thanks. A good amount of material!
qbet casino online new online casinos online casinos for android
You actually revealed that very well!
spinago online casino australia [url=https://usacasinomaster.com/#]casino online no deposit[/url] gold rush online casino
With thanks. Numerous info!
ace333 online casino [url=https://luckyusaplay.com/#]us online casinos with rtg software[/url] bally’s pa online casino
Cheers. Great stuff.
barstool sportsbook online casino no deposit online casino usa best of online casino
You actually expressed it effectively.
new online pa casinos social casino online miami dice online casino
Amazing loads of very good info.
casino online dinheiro real pix live casino online 1xbet casino online uruguay
Kudos! I value this.
novГ© ДЌeskГ© casino online [url=https://usaplayerscasino.com/#]online casino reviews[/url] casino online gratis ganhar dinheiro
Fine data. Appreciate it!
gta online casino time trial [url=https://usagamblinghub.com/#]online casino list[/url] best online casinos for denmark
Terrific data, Thanks!
gibraltar online casino no deposit online casinos no scam online casino
Terrific posts, Thanks a lot.
australian online casino that accepts paypal [url=https://usacasinomaster.com/#]best online casino bonuses[/url] skycity online casino no deposit bonus
Nicely put, Regards.
online casino immediate payout best casino online best online casino in us
Thanks, Helpful stuff.
online casinos in virginia social casinos online casino solitaire online
Thank you, I value it!
casino nj online best online casinos usa ac casino online no deposit bonus codes
Fantastic data. Regards!
thunderstruck 2 online casino [url=https://usaplayerscasino.com/#]online casinos with no deposit bonus[/url] online casino river monster casino
Wow plenty of fantastic knowledge!
phl96 online casino register free casino online online casino washington state
Very good posts. Kudos!
online casinos in deutschland [url=https://usagamblinghub.com/#]new usa online casinos real money[/url] new zealand casino online
Terrific content. Cheers!
betrivers online casino review michigan casino online online casino uk not on gamstop
Thank you! Lots of stuff!
redkings online casino review online casino bonuses best online casino for payouts
Wonderful information. Kudos.
best georgian online casino sites [url=https://luckyusaplay.com/#]best paying online casino[/url] mohegan online casino ct
Really many of very good data!
draftkings online casino pennsylvania live online casino holland casino poker online
Many thanks, I enjoy it.
mi online casino apps deposit bonus casino online online casino real money free play no deposit bonus
With thanks. I enjoy it!
bahraini online casino [url=https://uscasinoguides.com/#]best casino online[/url] top payout casinos online
Nicely put, Regards.
online casinos no deposit free play [url=https://usaplayerscasino.com/#]online casino no deposit bonus codes[/url] cГіdigos para casinos online
Many thanks. I enjoy it!
royal online casino new online casino no deposit bonus usa online casinos with no deposit free spins
Thanks. Plenty of information.
no deposit bonus codes for us online casinos [url=https://usagamblinghub.com/#]best paying online casino[/url] luckster online casino
Perfectly spoken indeed! !
list of online casinos for usa players online casino in usa easy win online casino
Terrific posts, Many thanks!
hyper online casino [url=https://usacasinomaster.com/#]best usa online casino[/url] what is online casino bonus
Wow a good deal of great material!
gta 5 online casino money glitch live dealer online casino prepaid credit card online casino
You’ve made your stand pretty effectively!.
hack online casino games [url=https://luckyusaplay.com/#]us online casino[/url] all new online casinos accepting south africa
Terrific information. Appreciate it!
1 casino online online casino reviews caesar palace online casino no deposit bonus
Wonderful data. Thanks!
tennessee online casino live casino online high roller online casino download for android
Thank you! Excellent stuff!
empire casino online login [url=https://uscasinoguides.com/#]play casino online free[/url] online casino oesterreich
You actually explained it well.
lady luck online casino no deposit bonus codes 2022 online casino bonuses nya online casino 2017
You stated it wonderfully!
ten online casino [url=https://usaplayerscasino.com/#]usa online casinos[/url] best diners club casinos online
Helpful forum posts. Thanks a lot!
payment gateway for online casino free casino online online casino odds
You reported it very well!
best online casino tennessee [url=https://usagamblinghub.com/#]social casino online[/url] real money online casino minnesota no deposit bonus
Appreciate it! Numerous data.
online casino utan spelgräns [url=https://usacasinomaster.com/#]game casino online free[/url] casino online 10 euro bonus
Great advice. Many thanks.
monopoly online casino casino online usa watch casino movie online free
Great write ups. Thanks a lot.
gta online diamond casino heist bugstars [url=https://luckyusaplay.com/#]online usa casino[/url] online casinos that
Whoa a lot of superb advice!
mohegan sun casino online real money fastest payout online casino online casino vermont
Cheers. Excellent stuff!
no limit casino online top 10 online casino new jersey online casino list
Great posts. Many thanks.
blackjack ballroom casino play baccarat online for money online casino bonuses newest online casinos for australian players
Cheers. Loads of information!
free online vegas casino games [url=https://usacasinomaster.com/#]new online casino no deposit bonus[/url] online casinos south carolina
Kudos. Helpful information.
bandar casino online deposit 50 ribu brand new online casinos usa no deposit bonus online casino promotions pa
You have made your point quite nicely!.
aces casino online online casino free play wild casino online
Perfectly spoken really! !
real casino slot online [url=https://usaplayerscasino.com/#]casinos online usa[/url] casinos online que regalan un deposito inicial para jugar
Fantastic forum posts. Thanks a lot!
juwa 777 online casino login download for android ios online casino free play no deposit online best casino games
With thanks. Loads of info!
online casino offshore usa online casino sands of the caribbean online casino
Superb knowledge. Appreciate it!
harrahs casino online gaming [url=https://usacasinomaster.com/#]new usa online casinos with no deposit bonuses[/url] betmgm online casino michigan login
Regards! I like it!
privГ© city online casino social casinos online diamond casino gta online location
Truly lots of terrific knowledge!
10 best online casinos [url=https://luckyusaplay.com/#]best online casino reddit no deposit bonus[/url] online casinos that accept discover
Thank you. I value it.
free online casino slot machine games no download no registration online casino free bonus no deposit ballys pa online casino
You actually suggested it terrifically!
panda master online casino [url=https://usaplayerscasino.com/#]free casino games online[/url] robert downey jr online casino app
With thanks, I like it!
new online social casino top 10 online casinos amex accepted online casinos
Nicely put, Thanks!
online casino faq deposit bonus casino online king online casino
Wow plenty of amazing data!
national casino online [url=https://usacasinomaster.com/#]casino usa online[/url] online casino new member bonus
Tips certainly applied!!
online casino mit echtgeld startguthaben ohne einzahlung 2021 [url=https://luckyusaplay.com/#]casino online casino[/url] is it safe to play online casino
Wow plenty of beneficial advice.
jackpot party casino online free https://uscasinoguides.com/tennis-betting/ casino royale online facebook
Truly lots of beneficial material!
casinos online que si pagan https://usaplayerscasino.com/poker-apps/ casino usa online no deposit
You actually suggested that fantastically!
top gaming1 online casino sites https://usacasinomaster.com/poker-apps/ coral online casino reviews
Nicely put. Regards.
spree casino online https://usaplayerscasino.com/legit-casinos/ best online casino nz real money
You said it adequately.!
jogos casino online gratis slot machines https://uscasinoguides.com/virginia-casinos/ best kiwi online casinos
Good tips. With thanks.
which online casinos pay real money https://usaplayerscasino.com/video-poker/ online free casino games
Very good data. Kudos.
bally online casino pa https://usaplayerscasino.com/slotland-review/ unibet casino online nj
Nicely put. Many thanks.
how to start an online crypto casino https://usacasinomaster.com/no-deposit-casinos/ rating online casino
You actually explained this fantastically.
online ontario casinos https://usacasinomaster.com/fast-payout/ all casino access points gta online
You expressed that effectively!
review online casino in usa https://usacasinomaster.com/real-money-bingo/ agen casino online maxbet
Kudos! Ample info!
hard rock social online casino https://usagamblinghub.com/slotocash-review/ casino nl online
Nicely put. Thanks!
mgm grand pa online casino https://luckyusaplay.com/california-casinos/ south african online casino free bonus
With thanks, I value this.
como se juega en casinos online https://usacasinomaster.com/massachusetts-casinos/ apuestas mejores casinos online
This is nicely expressed. .
best online live casino australia https://usaplayerscasino.com/ohio-casinos/ gta online pc casino glitch
This is nicely put. .
top online casino california https://usagamblinghub.com/ignition-review/ 7sultans online casino no deposit
You revealed this wonderfully.
casino games online no deposit bonus https://usaplayerscasino.com/minnesota-casinos/ real money usa online casino
Wonderful data. Thanks!
what is the best online casino game to play https://usagamblinghub.com/indiana-casinos/ online casino promotions uk
You suggested this perfectly.
canadian online casino zodiac https://usagamblinghub.com/virginia-casinos/ online casinos that accept vanilla prepaid mastercard
Very well expressed really. .
top 100 us online casinos https://usacasinomaster.com/nba-betting/ online casino programming
You’ve made the point!
https://usagamblinghub.com/poker-games/
Kudos. Good information!
https://uscasinoguides.com
ZaVdiGneWP
oLeQjIEDfzhY
deflazacort online order – buy calcort online cheap brimonidine eye drops
imusporin without prescription – purchase imusporin eye drops buy colchicine online
hytrin usa – purchase tamsulosin generic order priligy 60mg pill
finax pill – alfuzosin usa alfuzosin drug
norfloxacin cost – purchase confido for sale cheap confido generic
order generic lasuna – buy lasuna generic where to buy himcolin without a prescription
buy gasex pills – purchase gasex without prescription order diabecon
buy atorlip pills for sale – order nebivolol 20mg pills nebivolol canada
buy tenormin medication – betapace buy online buy generic carvedilol
order verapamil online – buy tenoretic pill tenoretic online order
arava where to buy – calcium carbonate pills buy generic cartidin over the counter
ascorbic acid drug – buy isordil for sale buy prochlorperazine for sale
buy ondansetron generic – generic ondansetron requip drug
buy cyclobenzaprine pills – buy generic olanzapine for sale order enalapril 10mg pills
cyclophosphamide for sale – cyclophosphamide canada cost trimetazidine
aldactone 100mg canada – order spironolactone without prescription revia 50mg sale
cheap disopyramide phosphate – cheap norpace pills buy thorazine 50 mg sale
order depakote 500mg – aggrenox generic topamax 200mg us
Very engaging and funny! For more information, visit: EXPLORE FURTHER. Let’s chat!
hydrea online buy – how to get crixivan without a prescription order methocarbamol 500mg online cheap
nootropil 800mg ca – sustiva 10mg canada order sinemet 20mg online
piroxicam for sale – rivastigmine 6mg canada rivastigmine 3mg drug
buy etodolac – buy cilostazol generic pletal online order
vasotec 5mg usa – buy doxazosin medication generic xalatan
pill dramamine 50 mg – cost risedronate 35 mg order actonel 35 mg without prescription
order forxiga generic – buy doxepin 25mg precose 25mg canada
fulvicin 250 mg us – lopid drug gemfibrozil medication
cotrimoxazole 480mg pill – buy keppra pills for sale buy tobrex for sale
brand zovirax – eukroma generic dydrogesterone 10mg uk
order dulcolax generic – dulcolax 5 mg ca order liv52 sale
rabeprazole pill – cost aciphex 20mg domperidone for sale online
florinef abroad – esomeprazole strange prevacid pills hero
biaxin experiment – mesalamine pills picture cytotec pills delicate
promethazine gallop – promethazine fence promethazine prefer
ascorbic acid either – ascorbic acid sideway ascorbic acid shrug
claritin pills putt – claritin dinner claritin pills exercise
priligy list – dapoxetine child priligy kindness
valacyclovir online area – valtrex pills vital valacyclovir snore
loratadine companion – loratadine medication greet claritin pills handsome
prostatitis pills gate – prostatitis medications miss pills for treat prostatitis tumble
inhalers for asthma pink – asthma medication scene asthma medication golden
dapoxetine conclusion – dapoxetine thud cialis with dapoxetine fork
cialis soft tabs pills bark – levitra soft online let viagra oral jelly spin
cialis soft tabs online bomb – viagra oral jelly online unicorn viagra oral jelly better
brand cialis shutter – viagra soft tabs probable penisole wire
cenforce perhaps – cialis no prescription brand viagra pills heaven
priligy chase – cialis with dapoxetine sum cialis with dapoxetine cheek
viagra professional twelve – levitra oral jelly exact levitra oral jelly online dinner
OCgsiWUNk
simvastatin onto – lopid over the counter lipitor climb
how to buy nitroglycerin – where can i buy valsartan order diovan 80mg
microzide us – buy zebeta 5mg pills bisoprolol cost
metoprolol 50mg pill – order nifedipine generic order generic nifedipine
oral digoxin – buy calan 240mg pills buy furosemide generic
famciclovir 500mg ca – cost famvir 250mg valcivir 1000mg generic
order generic ketoconazole 200mg – lotrisone buy online sporanox medication
UOqtDmjMgEaNdKA
terbinafine cost – order diflucan 200mg generic grifulvin v for sale online
cheap prandin 1mg – buy empagliflozin 10mg for sale jardiance 10mg generic
micronase 5mg cost – order actos purchase forxiga for sale
buy clarinex without prescription – desloratadine ca ventolin generic
how to get methylprednisolone without a prescription – methylprednisolone cost in usa buy astelin generic
albuterol cheap – buy promethazine 25mg pill buy theo-24 Cr 400 mg pills
order cleocin 300mg pill – cheap vantin chloromycetin pills
buy azithromycin generic – tindamax order online cost ciprofloxacin 500mg
purchase amoxil generic – cefuroxime for sale online order cipro
buy augmentin 625mg online – acillin drug order ciprofloxacin for sale
buy atarax 10mg pill – buy escitalopram 20mg pill buy amitriptyline 25mg
anafranil 50mg oral – buy paxil tablets sinequan 75mg sale
buy quetiapine sale – buy zoloft paypal eskalith without prescription
clozapine uk – how to buy clozaril buy famotidine for sale
zidovudine pills – buy generic glycomet online generic allopurinol 100mg
glucophage canada – brand glucophage order lincomycin 500 mg generic
buy furosemide tablets – order prograf 1mg generic capoten 120mg for sale
flagyl 400mg ca – cleocin 300mg cheap azithromycin 250mg cheap
valacyclovir 500mg cheap – cost valtrex 1000mg order zovirax generic
buy ivermectin 12mg – cefixime 100mg pills buy tetracycline medication
flagyl 200mg usa – purchase amoxicillin without prescription brand zithromax
ciprofloxacin 500 mg sale – buy generic amoxicillin
order erythromycin online
ciprofloxacin pills – bactrim 480mg uk augmentin 1000mg pill
cipro tablet – cephalexin price buy amoxiclav without prescription
lipitor 20mg uk order lipitor for sale atorvastatin pills
zqrSpaMTIjEevfHw
iIECgyne
请问测序那一步,具体怎么做啊,测序公司放假,那我是把基因组放在什么温度还是等他们上班再提基因组?谢谢!