在分子机制的研究中,蛋白与蛋白之间的互作可以称得上是非常经典实验。在进行蛋白功能研究时,寻找蛋白质之间的互作是比不缺少的一步。那么如何查询与预测蛋白质之间的互作呢?下面介绍一个预测蛋白质之间互作的网站-STRING,网址:http://www.string-db.org。
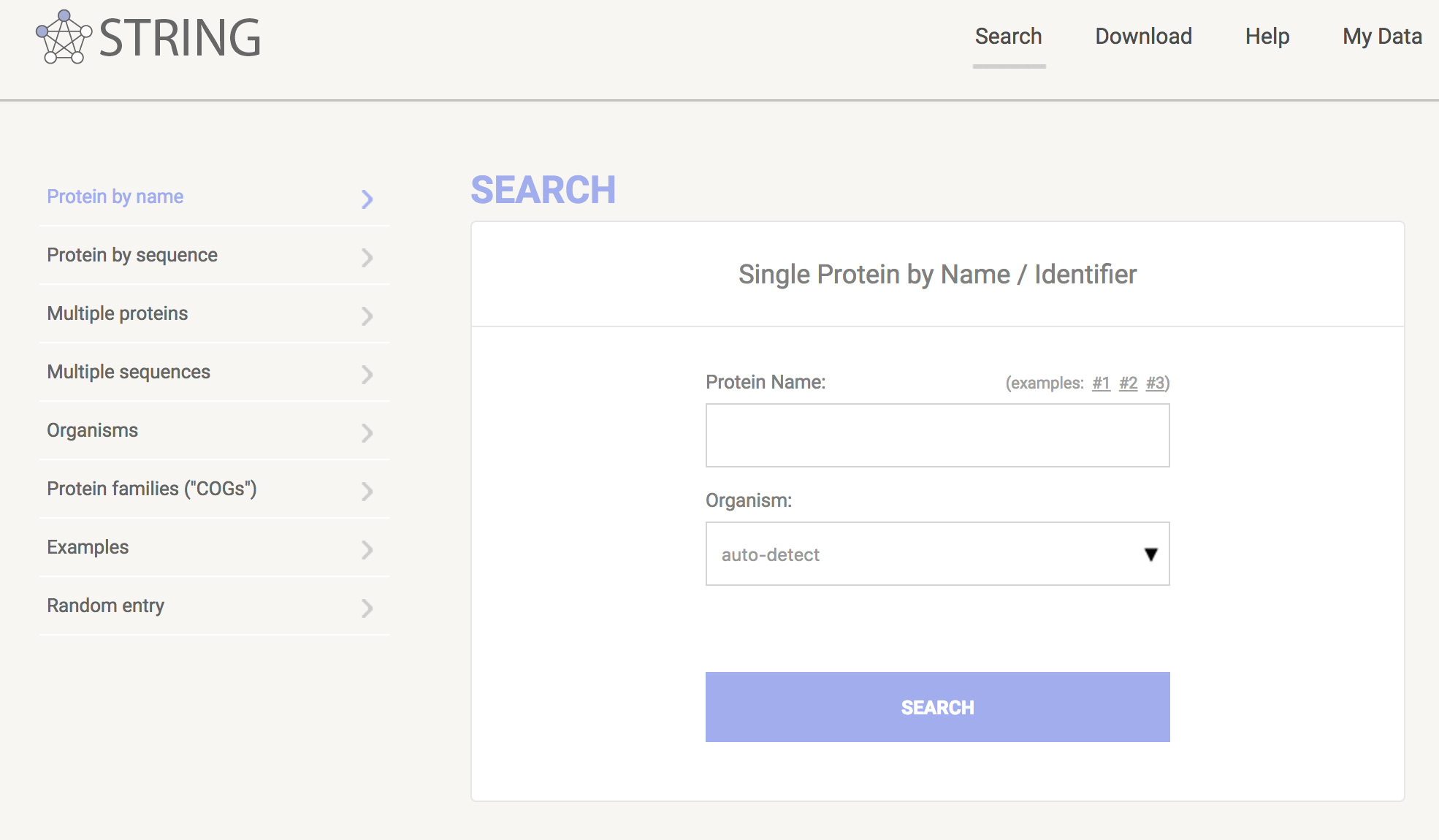
打开主页,在主页左侧栏显示搜索方式。比如输入一种蛋白质的名称或者序列,寻找与蛋白互作的其他蛋白;或者同时输入几种蛋白#A、#B、#C,搜索他们三者之间可能互作关系。当然,你也可以输入蛋白质家族进行查询。
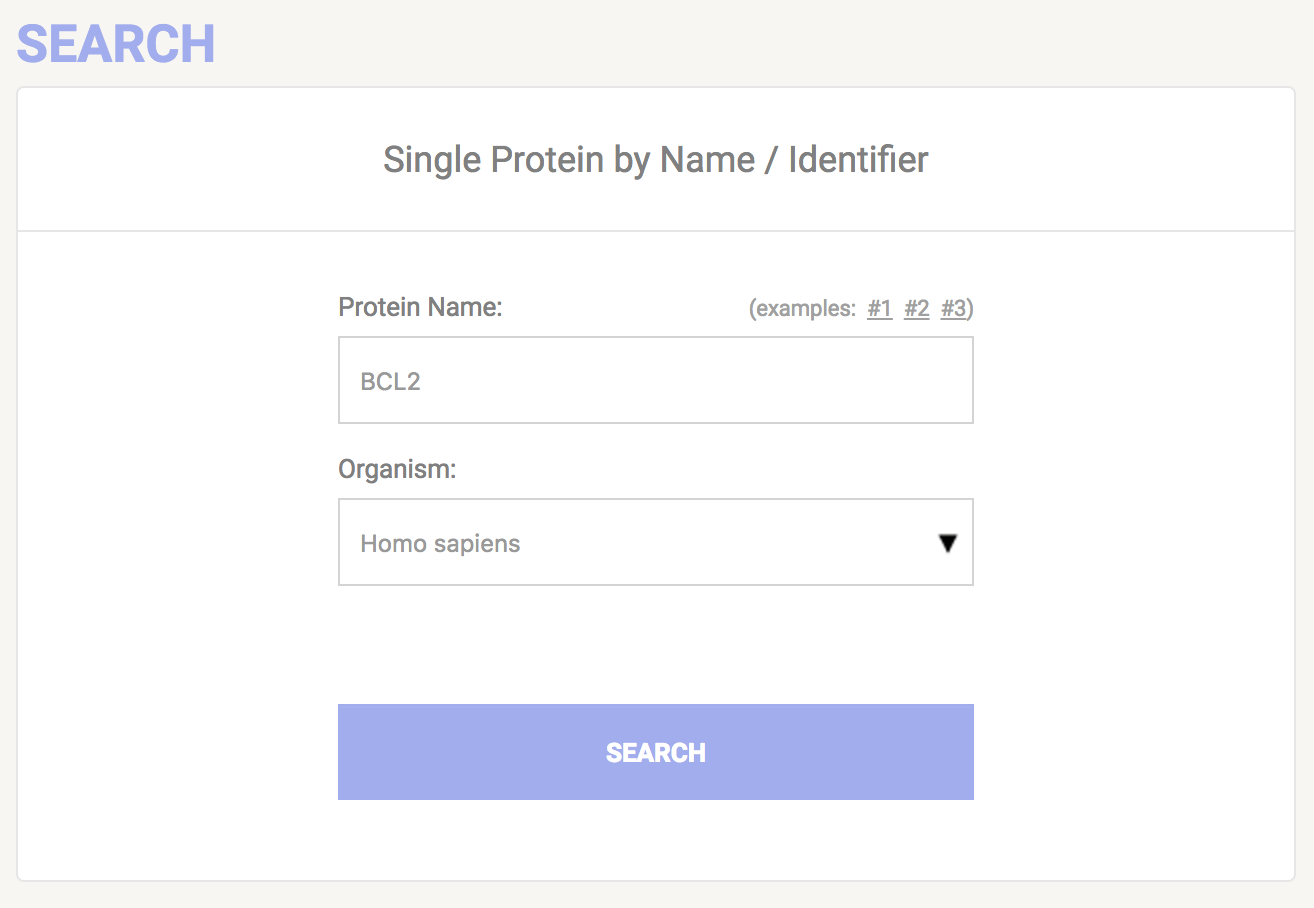
举个栗子,在搜索栏输进基因名称:BCL2,然后选择想要研究的种属,点击search。
首先看到BCL2与其他蛋白质之间的互作网络。可以通过点击蛋白名称的小球,比如TP53,来查看该TP53蛋白的基本介绍和所包含功能域的三维结构。
连接小球之间线条的颜色,表示蛋白之间互作的可信程度:蓝色是从专业数据库获得;紫色表示有相关实验证实;绿色呢,可能提示是基因位置临近蛋白……点击任意一条线条,可以看到两种蛋白之间互作的详细情况。
显示结果太复杂,看的眼花缭乱?那可以通过限定显示内容来解决。点击Settings,可以选择只搜索数据库内容或者实验证实的互作关系。
蛋白质互作预测网站旨在提供蛋白质互作的已经验证或者推测可能的互作,提供一种参考,接下来的任务是通过实验来证实,比如经典的蛋白互作实验包括:酵母双杂交实验,免疫共沉淀实验,GST-pulldown实验。











Its like you read my mind! You seem to know so much approximately this,
like you wrote the e-book in it or something. I think that you simply can do with some p.c.
to force the message house a bit, but other than that, this is
great blog. A fantastic read. I will definitely
be back.
my web site: vpn code 2024
excellent issues altogether, you just received a new reader.
What may you suggest about your post that you just made a few days in the
past? Any sure?
Here is my website vpn coupon code 2024
我记得Uniprot里面查询蛋白功能的时候,会有一些相互作用的数据库链接的