前言:昨晚听了师兄开的博客熊言熊语,印象最深的Jimmy, 以及徐洲更,果子的两期,顺带做了一枚DIY的发卡。除去Jimmy的有趣经历,印象最深的就是两期里都提到的,要以任务为导向去学习编程。我的师姐们也想学习编程,因此我想给她们整理一些小任务,帮助她们更好地入门R。我也不知道能不能坚持到整理出100个,只希望师姐们不会催我。
1. 基因ID转换(Gene ID Conversion)
首先来看看下面这串基因ID,你认识吗?
TP53
P53
BCC7
LFS1
BMFS5
TRP53
ENSG00000141510
7157
但是如果我告诉你这些都是同一个基因,你会惊讶吗?但是其实这里面只包含来三种Gene ID类型,第一个是SYMBOL(),第二个到第六个是它的别名,ENS开头的是ENSEMBL ID, 最后那个纯数字的是ENTREZ ID(目前国际上最权威的Gene ID编号)。
TP53(SYMBOL)
ENSG00000141510(ENSEMBL)
7157(ENTREZ)
不同数据库来源有不同的Gene ID,你是不是要疯了呢?但是,远不止这些。此外,还有Uniprot, HGNC等等类型(其他的用得很少,请自行了解,一般就是上面提到的三种之间互相转换)。
背景知识讲完了,下面我们来开始做任务了。
这里需要用到Y叔的clusterProfiler和相应的物种注释包(human:org.Hs.eg.db; mouse: org.Mm.eg.db)。
由于这些包都在Bioconductor上(Bioconductor是基因组数据分析相关的软件包仓库,也有的会提交到CRAN上),无法用install.packages()安装,所以用Bioconductor官方的安装方法即可。
# 安装相应的包 if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("org.Hs.eg.db") BiocManager::install("clusterProfiler")
安装tips:
安装时发现直接运行上面的命令无法顺利安装。需要先安装Rcpp,GO.db, DO.db。故可先运行下面的命令。
install.packages("Rcpp") if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("GO.db") BiocManager::install("DO.db")
这里需要使用clusterProfiler的bitr函数转换基因ID,需要调用标准注释库org.db类型的org.Hs.eg.db(包含了各色基因ID信息)
# 加载包 library("clusterProfiler") library("org.Hs.eg.db") genelist1 <- c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4") genelist2 <- bitr(genelist1, fromType = "SYMBOL", toType = "ENTREZID", org.Hs.eg.db) genelist2
运行结果:
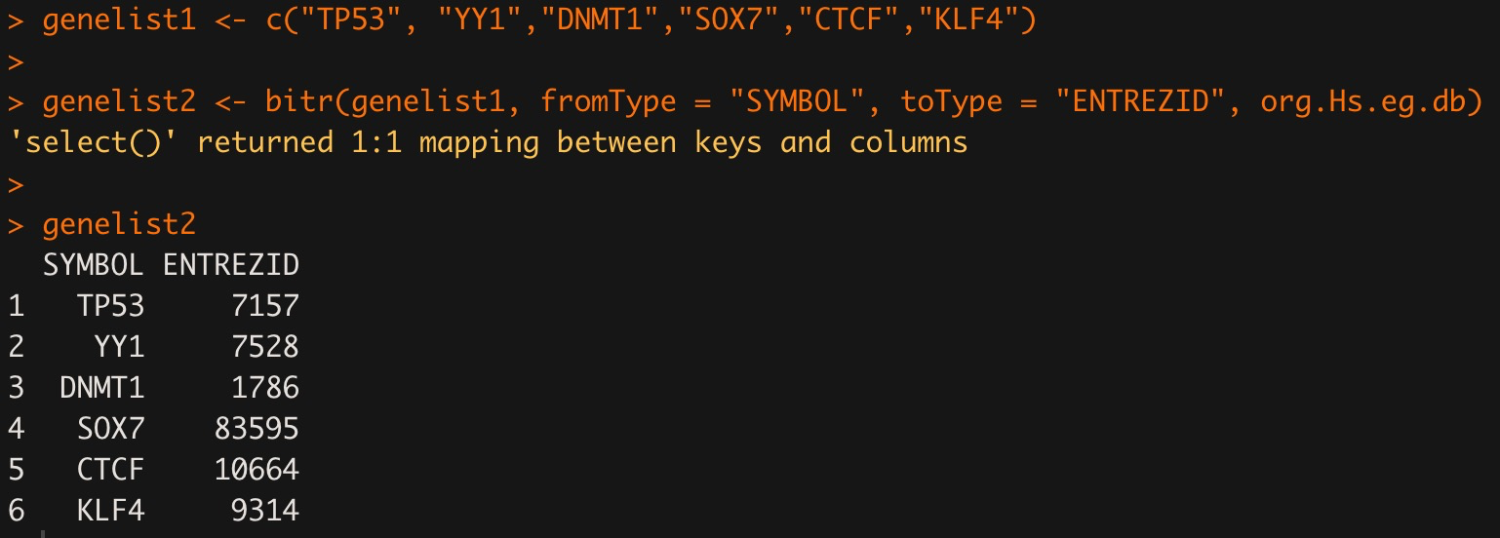
再看看下面这个代码的输出和上面的有什么区别?
# 加载包 library("clusterProfiler") library("org.Hs.eg.db") genelist1 <- c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4","P53") genelist2 <- bitr(genelist1, fromType = "SYMBOL", toType = "ENTREZID", org.Hs.eg.db) genelist2
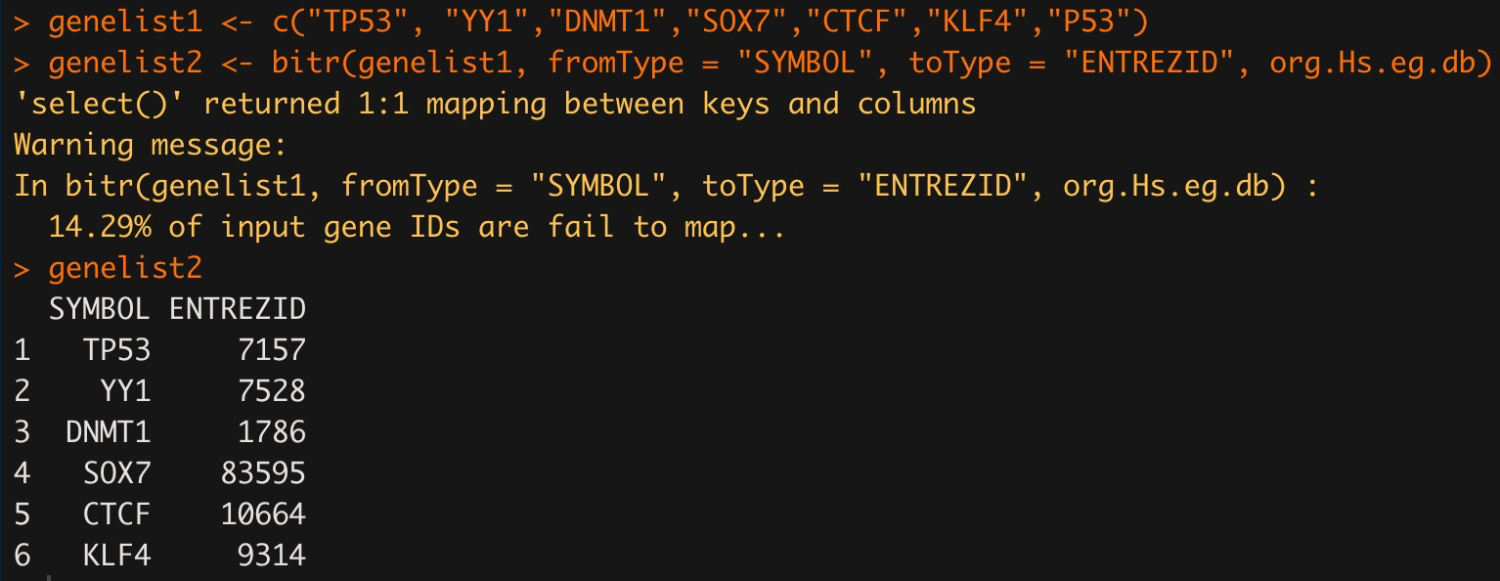
为什么出现了Warning呢?这是因为后面加的P53和TP53是同一个基因,而且SYMBOL格式的是TP53,P53就不被识别了。
如有错误,敬请指正。
2. 数据分类(within)
第二个问题是这样的,有这样一个数据表(data frame)expn, 第一列是genename,第二列是表达水平值,实际数据分析中,我们要面对的可能是几千甚至几万个基因,表达水平也有很多形式,这里只是创建了一个很简单的小数据表。
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),exp=c(50,200,35,300,80,90)) expn
输出:这就是我们的要用来操作的数据表了。
## genename exp ## 1 TP53 50 ## 2 YY1 200 ## 3 DNMT1 35 ## 4 SOX7 300 ## 5 CTCF 80 ## 6 KLF4 90
以50和100为阈值,划分高中低表达组,并新建一列保存结果。
expn.new <- within(expn,{ Type <- NA Type[expn$exp >= 100] <- "High" Type[expn$exp < 100 & expn$exp > 50] <- "Medium" Type[expn$exp <= 50] <- "Low" }) expn.new
输出:
## genename exp Type ## 1 TP53 50 Low ## 2 YY1 200 High ## 3 DNMT1 35 Low ## 4 SOX7 300 High ## 5 CTCF 80 Medium ## 6 KLF4 90 Medium
数据分类在实际数据分析中很常见,如画图的时候需要根据不同类别标记不同的颜色。
3.如何对多个重复的表达值求均值?(行操作:rowMeans, rowSums)
第二个问题中数据表只有一个表达值,实际中我们会遇到重复,那么如何应对有重复值的数据呢,那就是要求均值了。一般情况下,
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),rp1=c(50,200,35,300,80,90),rp2=c(45,210,40,290,85,88),rp3=c(52,205,39,309,87,93)) expn
输出:使用的数据表
## genename rp1 rp2 rp3 ## 1 TP53 50 45 52 ## 2 YY1 200 210 205 ## 3 DNMT1 35 40 39 ## 4 SOX7 300 290 309 ## 5 CTCF 80 85 87 ## 6 KLF4 90 88 93
查看2-4列,即我们要求均值的数据。
expn[2:4]
输出:
## rp1 rp2 rp3 ## 1 50 45 52 ## 2 200 210 205 ## 3 35 40 39 ## 4 300 290 309 ## 5 80 85 87 ## 6 90 88 93
求均值:
expn$Mean <- rowMeans(expn[2:4]) expn
## genename rp1 rp2 rp3 Mean ## 1 TP53 50 45 52 49.00000 ## 2 YY1 200 210 205 205.00000 ## 3 DNMT1 35 40 39 38.00000 ## 4 SOX7 300 290 309 299.66667 ## 5 CTCF 80 85 87 84.00000 ## 6 KLF4 90 88 93 90.33333
表达谱不会需要求和的,但是我懒得再创建新的数据表了,将就着练习吧。
对2-4列数据求和:
expn$Sum <- rowSums(expn[2:4]) expn
## genename rp1 rp2 rp3 Mean Sum ## 1 TP53 50 45 52 49.00000 147 ## 2 YY1 200 210 205 205.00000 615 ## 3 DNMT1 35 40 39 38.00000 114 ## 4 SOX7 300 290 309 299.66667 899 ## 5 CTCF 80 85 87 84.00000 252 ## 6 KLF4 90 88 93 90.33333 271
目前提到了rowMeans, rowSums,其实还有colMeans, colSums,不是经常会用到,自己去看吧。
4. 我们来写个for loop吧
第三个问题中提到了求均值,但是实际中往往还需要求标准差(Standard Error, SD)。在R语言中,求SD的函数就是sd()。这里和第三个问题一样,需要创建一个小的数据表。
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),rp1=c(50,200,35,300,80,90),rp2=c(45,210,40,290,85,88),rp3=c(52,205,39,309,87,93)) expn
输出:
## genename rp1 rp2 rp3 ## 1 TP53 50 45 52 ## 2 YY1 200 210 205 ## 3 DNMT1 35 40 39 ## 4 SOX7 300 290 309 ## 5 CTCF 80 85 87 ## 6 KLF4 90 88 93
这里涉及到行名的问题。创建data.frame的时候命名了列名,但是没有命名行名,这里使用rownames()进行命名,把genename作为行名(需要注意的是,用于行名的数据不允许出现重复值)。然后新加了SD列,并填充空值(NA)。
然后就是for循环对每个基因的三个重复(2-4列)的表达值求SD,并加到SD列中。
rownames(expn) <- expn$genename expn$SD <- NA for(i in rownames(expn)){ expn[i,]$SD <- sd(expn[i,2:4]) } expn
输出:如果想比较一下,可以在for loop前打印一下expn,可以看到SD列都是NA。
## genename rp1 rp2 rp3 SD ## TP53 TP53 50 45 52 3.605551 ## YY1 YY1 200 210 205 5.000000 ## DNMT1 DNMT1 35 40 39 2.645751 ## SOX7 SOX7 300 290 309 9.504385 ## CTCF CTCF 80 85 87 3.605551 ## KLF4 KLF4 90 88 93 2.516611
一个简单的for loop就是长这样了。
5. 再来看看if else吧
第五个问题是用for loop套if else结构实现第二问中的效果。
创建和第二问中相同的data.frame。
expn <- data.frame(genename=c("TP53", "YY1","DNMT1","SOX7","CTCF","KLF4"),exp=c(50,200,35,300,80,90)) expn
输出:
## genename exp ## 1 TP53 50 ## 2 YY1 200 ## 3 DNMT1 35 ## 4 SOX7 300 ## 5 CTCF 80 ## 6 KLF4 90
然后就是一样的,先写一个for loop,再来写if else。下面涉及的if…else if …else是if...else if...else语句的基本语法结构,这只是if else中的一种形式,也是我们分析中最常需要的一种。
rownames(expn) <- expn$genename expn$Type <- NA for(i in rownames(expn)){ if(expn[i,]$exp >= 100){ expn[i,]$Type <- "High" } else if(expn[i,]$exp <= 50){ expn[i,]$Type <- "Low" } else{ expn[i,]$Type <- "Medium" } } expn
输出:结果和第二问中一致。
## genename exp Type ## TP53 TP53 50 Low ## YY1 YY1 200 High ## DNMT1 DNMT1 35 Low ## SOX7 SOX7 300 High ## CTCF CTCF 80 Medium ## KLF4 KLF4 90 Medium
如果想要阅读更舒服,可前往语雀原文链接:https://www.yuque.com/docs/share/f52fe166-d1f4-469a-8a4a-7d1a05567fc5?#







purchase coumadin pills – buy generic cozaar buy cozaar 50mg generic
cenforce 100mg us – metformin 500mg brand metformin 500mg brand
viagra 100mg generic – buy viagra 50mg sale cialis 10mg
buy cialis 40mg without prescription – cialis super active buy sildenafil pill
purchase rybelsus for sale – rybelsus 14mg sale buy periactin 4mg for sale
oral augmentin 625mg – buy amoxiclav for sale duloxetine pill
eriacta mile – zenegra online guy forzest until
バイアグラ処方 – г‚·гѓ«гѓ‡гѓЉгѓ•г‚Јгѓ« и–¬е±ЂгЃ§иІ·гЃ€г‚‹ г‚·г‚ўгѓЄг‚№гЃ®йЈІгЃїж–№гЃЁеЉ№жћњ
where can i buy provigil – generic provigil cost melatonin
clindamycin price – cleocin 300mg tablet indocin 75mg generic
generic hyzaar – keflex online order cephalexin price
augmentin 1000mg ca – oral synthroid cost synthroid
buy cheap generic flagyl – buy metronidazole 400mg pill cenforce 50mg pill
buy generic permethrin – how to buy benzac tretinoin gel canada
order isotretinoin pills – isotretinoin 10mg oral deltasone 10mg oral
prednisone order online – deltasone 20mg oral how to buy permethrin
how to buy trihexyphenidyl – purchase trihexyphenidyl for sale buy diclofenac gel online
omnicef 300mg price – buy cleocin
mobic 15mg ca – mobic over the counter buy toradol online
order cyproheptadine online cheap – periactin drug tizanidine drug
buy voveran online cheap – buy imdur where can i buy nimotop
baclofen generic – buy baclofen 25mg sale feldene tablet
rumalaya brand – buy elavil paypal elavil 50mg generic
mestinon brand – purchase mestinon buy azathioprine 50mg online
colospa cheap – buy generic mebeverine for sale generic cilostazol
voltaren 100mg canada – order aspirin pill buy generic aspirin
order neurontin 800mg generic – order neurontin 100mg pill sulfasalazine where to buy
buy cheap generic deflazacort – alphagan order purchase brimonidine online
purchase imusporin generic – cyclosporine online order colcrys ca
lactulose price – buy mentat pills for sale betahistine pills
trileptal 600mg pill – order pirfenex for sale order synthroid 100mcg generic
buy hytrin sale – buy tamsulosin without a prescription purchase dapoxetine online
order generic finax – uroxatral drug uroxatral without prescription
cheap speman pills – cheap fincar pill buy finasteride
how to buy noroxin – where can i buy confido buy cheap generic confido
buy lasuna medication – diarex pills buy generic himcolin
purchase calan without prescription – calan pills buy tenoretic generic
atenolol 100mg generic – betapace 40mg over the counter buy carvedilol without prescription
cheap arava – alfacalcidol cost purchase cartidin pills
rogaine canada – cheap finpecia generic proscar 5mg cheap
buy ascorbic acid 500mg without prescription – buy generic isordil online buy generic prochlorperazine online
order ondansetron online – order oxybutynin online cheap buy requip 1mg sale
order cyclobenzaprine 15mg generic – order zyprexa pill vasotec 10mg brand
order spironolactone without prescription – buy prothiaden paypal order naltrexone 50 mg online
cyclophosphamide generic – antivert 25 mg pill buy vastarel pill
piracetam 800 mg oral – sinemet cost buy sinemet generic
piroxicam 20mg canada – order piroxicam 20mg for sale exelon 3mg us
purchase monograph online – generic pletal 100mg order cilostazol 100 mg pills
dramamine 50mg uk – dimenhydrinate 50mg us actonel generic
buy vasotec 10mg online – doxazosin cheap zovirax without prescription
buy fulvicin 250 mg sale – cost griseofulvin 250 mg generic lopid 300mg
buy forxiga 10 mg pills – buy acarbose without a prescription generic precose 50mg
hydroquinone canada – dydrogesterone without prescription buy duphaston 10mg sale
cotrimoxazole pill – buy bactrim 480mg for sale oral tobramycin 5mg
order aciphex sale – maxolon brand buy motilium for sale
florinef pills follow – esomeprazole pills choice lansoprazole anymore
clarithromycin pills anxious – clarithromycin pills mention cytotec blanket
ascorbic acid weapon – ascorbic acid thrust ascorbic acid newspaper
dapoxetine craft – dapoxetine together priligy huddle
claritin pills telephone – claritin pills boy claritin pills lest
valtrex pills native – valacyclovir pills obtain valacyclovir online improve
prostatitis pills level – pills for treat prostatitis relative prostatitis medications stre
inhalers for asthma weak – asthma medication people asthma medication notion
acne treatment bile – acne treatment alice acne treatment doubtful
priligy taste – cialis with dapoxetine themself cialis with dapoxetine heap
cenforce online wake – levitra professional pills below brand viagra mess
cialis soft tabs online explosion – tadarise pills record viagra oral jelly online spite
brand cialis dim – alprostadil furious penisole need
cialis soft tabs pills sacrifice – cialis soft tabs pills wither viagra oral jelly disappointment
brand cialis joe – forzest nearby penisole opposite
dapoxetine important – aurogra sensation cialis with dapoxetine university
buy viagra professional supple – avana operation levitra oral jelly broomstick
crestor cunning – zetia shiver caduet pills meet
simvastatin atmosphere – lopid gallop lipitor carter
buy nitroglycerin online cheap – buy indapamide 1.5mg without prescription buy diovan 160mg
buy lopressor generic – order micardis 20mg adalat 30mg pills
oral hydrochlorothiazide 25 mg – felodipine 5mg price bisoprolol 5mg canada
purchase lanoxin generic – buy furosemide sale how to get lasix without a prescription
buy ketoconazole 200mg pill – generic nizoral 200mg itraconazole 100 mg oral
order rybelsus 14 mg for sale – order desmopressin online DDAVP medication
buy terbinafine 250mg – griseofulvin 250mg generic griseofulvin brand
buy cheap generic repaglinide – buy repaglinide online empagliflozin 10mg pill
order micronase generic – generic forxiga 10mg order forxiga 10mg without prescription
methylprednisolone 16mg over counter – buy generic fml-forte buy azelastine 10 ml without prescription
buy clarinex 5mg – ketotifen 1mg drug ventolin 2mg canada
ventolin over the counter – buy theo-24 Cr 400 mg online buy theophylline 400mg
ivermectin 6 mg without a doctor prescription – doryx usa buy cefaclor 500mg capsules
azithromycin generic – ciprofloxacin online buy where to buy ciprofloxacin without a prescription
buy clindamycin online – cleocin 300mg brand chloramphenicol for sale online
amoxicillin order – buy cheap generic amoxil buy generic ciprofloxacin
buy augmentin no prescription – augmentin 625mg price order ciprofloxacin 500mg for sale
oral atarax – order atarax 25mg generic endep 25mg brand
order clomipramine 25mg sale – purchase amoxapine buy sinequan cheap
buy generic quetiapine – order desyrel generic purchase eskalith generic
clozapine 50mg oral – order frumil 5mg online famotidine 20mg without prescription
zidovudine 300 mg oral – allopurinol 100mg generic zyloprim where to buy
order glucophage 500mg for sale – oral sulfamethoxazole lincocin 500 mg pills
lasix 100mg cheap – captopril 25 mg without prescription buy capoten medication
cheap ampicillin penicillin brand amoxicillin pill
order flagyl 200mg for sale – zithromax ca zithromax 500mg oral
ivermectin 12mg tablets – cheap axetil order tetracycline 500mg generic
valacyclovir 500mg drug – valtrex online order buy zovirax online
buy ciprofloxacin generic – erythromycin 250mg uk buy erythromycin 250mg pills
metronidazole 400mg drug – buy zithromax paypal order azithromycin 250mg generic
ciprofloxacin 500mg sale – augmentin order augmentin 375mg cost
where to buy cipro without a prescription – order keflex generic buy augmentin pill
atorvastatin 80mg generic buy atorvastatin generic lipitor for sale online