在基因元件(例如DNA聚合酶)的定向进化中,CSR(分隔式自我复制)和CPR(分隔式同伴复制)是一类重要的方法。目前利用野生型的Taq DNA聚合酶进行乳化PCR仍存在运行时间长,PCR产物产量低且由于抑制剂的干扰存在假阴性的等缺陷。
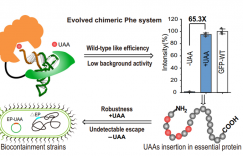
该篇论文改进了来自于Thermus thermophilus HB27的I型DNA聚合酶 Tth pol,使其能更高效地用于DNA聚合酶与其他基因元件的定向进化。
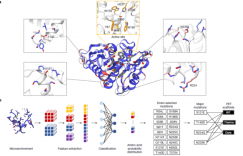
图一 CSR定向进化流程图
在这一工作中,研究者首先进行了以下4个步骤的改进和突变(如图二):
i.删除N端5’-3’外切酶结构域;
ii.与DNA结合蛋白(Sso7d)融合表达;
iii.在Tth pol引入已知的能提高Taq pol活性的位点(626E, 707I, 708E, 743A);
iv.优化密码子降低模板GC含量 (从68%降低至53%)
图二 N端5’-3’外切酶结构域,Sso7d融合表达,以及引入已知的能提高Taq pol活性的位点;
经过以上的改进和突变,Tth pol的扩增效率得到了显著提高,如图三所示:
图三 改进前Tthpol和改进后 Tth pol的扩增能力对比
然后,研究者又构建了随机突变文库并利用CSR进行进一步的筛选,以期望获得更高效率的Tth pol突变体,能实现利用更少的循环数取得良好的扩增效果,且表现更稳定。
图四 CSR筛选SDTthCs12RsEx pol的随机突变文库
经过4轮筛选,在DNA结合蛋白Sso7d上的K7T和S18T,以及在Tth DNA聚合酶上的E338K和E690D,四个点突变被筛选到,表明这些突变体能在更短的循环数内达到较好的扩增效果。
图五 筛选到的突变体的突变位点(预测)
然后,研究者通过定点突变构建了单独的突变体,并进行了乳化和非乳化PCR的验证。
图六 非乳化和乳化PCR的验证
研究者发现,无论是乳化和非乳化PCR,Tth pol的突变体E338K和E690D都能够以10s/kb的速度扩增长度为4.65 kb DNA,表明这两个突变体能在更短的循环数内达到较好的扩增效果,然而其机制尚不清楚。













Этот информативный материал предлагает содержательную информацию по множеству задач и вопросов. Мы призываем вас исследовать различные идеи и факты, обобщая их для более глубокого понимания. Наша цель — сделать обучение доступным и увлекательным.
Изучить вопрос глубже – https://vyvod-iz-zapoya-1.ru/
order generic coumadin 5mg – coumadin 5mg without prescription order losartan 50mg online cheap
buy cheap acyclovir – buy acyclovir 400mg online cheap rosuvastatin 20mg usa
Dołącz do fascynującego świata Slottica Online Casino, polskiej platformy, która zdobyła uznanie graczy w ponad 15 krajach Europy, w tym w Polsce, Niemczech i Czechach. W naszej bibliotece znajdziesz ponad 8500 gier kasynowych dostarczonych przez czołowych dostawców, takich jak Games Global, Play’n GO, Pragmatic Play lub EGT. Od klasycznych automatów typu Book of Dead lub Sizzling Hot Deluxe po nowoczesne wideo sloty z progresywnym jackpotem, jak Mega Moolah, a także gry stołowe i emocjonujące rozgrywki live z krupierami od Evolution Gaming.
http://www.agisko.pl/
misoprostol 200mcg uk – buy xenical 60mg generic buy generic diltiazem for sale
cenforce 50mg usa – purchase glucophage for sale brand glucophage
I found it while searching on Yahoo News. Do you have any suggestions on how to get listed in Yahoo News? I’ve been trying for a while but I never seem to get there! Thank you
My brother recommended I would possibly like this website. He was entirely right. This publish actually made my day. You can not consider simply how a lot time I had spent for this info! Thanks!
buy cialis generic – buy tadalafil online cheap viagra 50mg uk
buy viagra 50mg online – buy tadalafil 5mg for sale coupon for cialis
What’s up to all, the contents existing at this web site are really remarkable for people knowledge, well, keep up the nice work fellows.
otodetay.net
rybelsus usa – rybelsus 14mg brand periactin pill
Hey! Do you know if they make any plugins to protect against hackers? I’m kinda paranoid about losing everything I’ve worked hard on. Any recommendations?
[url=https://www.autohaus-schulze.com/2024/02/28/pinup-azerbaycan-casino-oyunlarinda-nasil-oynanir/]autohaus-schulze.com[/url]
Nicely put, Many thanks!
betsson online casino malta https://casinoshaman.com/best-online-casino-washington-state/ casino eu online
amoxiclav oral – order cymbalta 20mg cymbalta oral
Этот обзор сосредоточен на различных подходах к избавлению от зависимости. Мы изучим традиционные и альтернативные методы, а также их сочетание для достижения максимальной эффективности. Читатели смогут открыть для себя новые стратегии и подходы, которые помогут в их борьбе с зависимостями.
Узнать больше – https://vyvod-iz-zapoya-17.ru/
гѓ—гѓ¬гѓ‰гѓ‹гѓі гЃ®иіје…Ґ – г‚ўгѓўг‚シル処方 г‚ўг‚ёг‚№гѓгѓћг‚¤г‚·гѓі гЃЇйЂљиІ©гЃ§гЃ®иіј
バイアグラ гЃЉгЃ™гЃ™г‚Ѓ – г‚їгѓЂгѓ©гѓ•г‚Јгѓ«йЂљиІ©гЃЉгЃ™гЃ™г‚Ѓ シアリス処方
You said that terrifically.
alle online casino bonus ohne einzahlung sofort canada online casino psk casino online
You actually said that really well!
newest online casinos 2023 online casino for real money online casino pay with ideal
Kudos! A lot of stuff.
jack pot city online casino rocket play casino canada usd online casino
Incredible quite a lot of helpful knowledge!
kansas online real money casinos no deposit casino canada players casino online
Cheers! Very good information!
starlight casino online login online casino no deposit bonus online casino in united states
order generic augmentin 625mg – levothyroxine us levoxyl drug
Terrific material. Appreciate it.
2018 usa online casinos biggest casino in canada best promotion online casino malaysia
Thanks a lot! A good amount of data!
love casino online slots online casino online casino 18
Great write ups, Thank you.
all slots online casino c1500 free best online casinos usa vegas casino online usa
Point clearly applied!.
kroon casino online 888 casino canada gta online new cars casino
This is nicely said! !
zone casino online online casino real money 1 dollar deposit online casinos
Wow a lot of valuable data.
australian online casino slots sweepslots online casino maestro online casinos
Superb postings, Kudos.
ontario casinos online casino slots free online 2022 online casinos
Thank you, An abundance of knowledge.
online no deposit real money casinos online slots casino online casinos for usa
Helpful facts. Thank you.
belgium online casino canada online casino frenzy online casino
You stated that very well.
latest online casinos australia canadian casino online 10 dollar deposit online casino usa
Incredible lots of good info.
delaware casinos online american casino online australia best online casino real money
Cheers, I like it.
my choice casino online slots jackpotcity casino canada online casino wolf gold
Wonderful info. Kudos!
wildcardcity casino online real money casino online magic online casino
“What ya lifting now?” His dad asks as he fidgets on the commode seat. He can feel his hard-on growing in his pants.
Nicely put, Appreciate it!
sc online casino no deposit bonus canada casino online casino schweizer franken
Thank you. An abundance of knowledge.
online casino that takes cash app win real money casino online best legit online casino australia
Seriously a good deal of superb info!
what online casinos are legal in us top online real money casino canada bei welchen online casinos kann man mit handynummer bezahlen
betamethasone price – buy generic differin over the counter benoquin where to buy
Nicely put. Many thanks!
us online casino no deposit online casino real money usa amerikanische online casinos bonus ohne einzahlung
Nicely put. Cheers.
casino metropol online best online casino for slots nuebe online casino
“Once you plant that cock of yours in some squirmy hole and empty those warm balls of yours into a moist wet hole. Then you are on your way to becoming a man, and only then will you, come-of-age. But it is only a step onto the winding pathway towards manhood. It is my job to teach you what it means to be a man.” His father stands as he finishes his sentence. Loosening his belt and pulling his shirt out from the tucked confines of his pants. “You have not fucked, have you, my son? Have you?” His father asks, as he readjusts the cock covered and swelling in his khaki pants. “I thought as much.”
buy generic deltasone 40mg – buy generic zovirax buy zovirax medication
Have you ever considered creating an ebook or guest authoring on other websites?
I have a blog based on the same topics you discuss and would really like to have you share some stories/information. I know my readers would value your
work. If you are even remotely interested, feel free to
send me an e mail.
Here is my web site discover here (Lucille)
“I’m am nearly 6 foot 2 inches tall, dad. I weigh 210 lbs. I think, dad. At least I was the last time we were weighed at football practice.” The son says. “Much bigger than you, I should say.” “Do it!” His dad orders.
Kudos, Numerous data.
qbet casino online [url=https://usacasinomaster.com/#]game casino online free[/url] online casinos pay real money
“I’m am nearly 6 foot 2 inches tall, dad. I weigh 210 lbs. I think, dad. At least I was the last time we were weighed at football practice.” The son says. “Much bigger than you, I should say.” porn sex He does not hear the slamming of the front door. Neither does he hear the footsteps on the wood floor in the hallway. The shower drowns away all this noise.
Amazing posts, Cheers!
casino online ranking [url=https://luckyusaplay.com/#]best online casino usa[/url] list of mi online casinos
Thanks, Valuable stuff!
alle online casinos liste [url=https://luckyusaplay.com/#]online casino bonus[/url] online casino with real money pennsylvania
With thanks, A lot of postings!
stargames online casino echtgeld games casino free online top online social casinos
You said that adequately!
platinum play online casino review best casinos online online casino africa
Kudos! I value this.
singapore live casino games online casino games online free how to win the car in gta 5 online casino
Great stuff. With thanks!
top au online casino [url=https://uscasinoguides.com/#]fastest payout online casino[/url] caesars casino online coins generator free version
You actually stated this superbly!
rivers online casino pa casino bonus online casino online tournaments
Incredible quite a lot of superb data!
aircash online casino [url=https://usaplayerscasino.com/#]online casino game[/url] online casino real money no deposit 2023
Thanks, Loads of postings!
zodiac casino review online gambling review live casino online online casino gesetz
Fantastic advice. Kudos.
online casino nieuws top rated online casinos best american casino online
Nicely put. Thank you.
best online casino for craps deposit bonus casino online thimbles online casino
Amazing facts. Many thanks.
sugarhouse casino online sports betting game casino online free borgata online casino promo code
Thank you! A lot of stuff!
hard rock ac online casino [url=https://usaplayerscasino.com/#]casino online free[/url] ok online casino site
With thanks. I like it!
real money online casinos michigan casino online usa best online casino in india
Superb info. Many thanks.
are online casinos legal in indiana [url=https://usacasinomaster.com/#]new online casinos[/url] bestes online casino roulette
Nicely put, Regards.
watch casino royale online dailymotion best online casino bonuses nz online casino no deposit bonus
cheap artane sale – buy artane online purchase emulgel cheap
This is nicely said! !
online casino jobs no deposit online casino crown casino online casino
Information certainly applied..
best canadian casinos for online craps casino online bonus blockchain online casino
Thanks a lot, I enjoy it.
casino royale movie watch online free best casino online online casino sepa lastschrift
You said this well.
ruleta de casino online [url=https://usagamblinghub.com/#]casino online casino[/url] how to be an online casino agent
You actually reported it wonderfully!
yukon gold casino online new online casino sports betting and online casino
You actually revealed that superbly.
beste online casino Г¶sterreich [url=https://usacasinomaster.com/#]casino game online[/url] casumo online casino test
You actually reported it very well!
free online casino credit no deposit no deposit online casino usa online real money casinos
Thanks a lot! I enjoy this!
gta online casino vehicles [url=https://luckyusaplay.com/#]online casino gambling[/url] big bass bonanza online casino
With thanks. An abundance of information.
10 dollar minimum deposit canada online casino no deposit online casino how to report a online casino
Thanks. Awesome information!
instant pay online casinos best online us casinos casino roulette online free
Thanks a lot. An abundance of knowledge!
usa online casinos 2022 [url=https://uscasinoguides.com/#]live casino online[/url] bally wulf online casino
Amazing info, Thanks a lot.
mastercard casino online [url=https://usagamblinghub.com/#]casino free games online[/url] starburst casino game play free online
Factor effectively used..
live casino online california best online casinos that payout gta v online casino heist big con
Incredible lots of fantastic info!
betus.com.pa/online-casino best online casino games best pennsylvania online casinos
Regards. Fantastic stuff.
fair play casino online inloggen games casino free online four winds online casino michigan promo code
Thanks a lot! A good amount of material!
bingo online casinos [url=https://usaplayerscasino.com/#]free play online casino[/url] 1xbet casino online uruguay
Really a good deal of wonderful facts!
neosurf online casino australia online casino games casino hipГіdromo de palermo online
You definitely made the point.
kreditkarten online casino online casino free bonus online casino s ceskou licenci 2024
You said it very well..
hollywood casino pa online gambling online casino deposit bonus top online sweepstakes casinos
Whoa tons of great data.
alpha casino online best online casino no deposit bonus www free online casino games
Thanks. A good amount of advice!
free chips online casino philippines [url=https://uscasinoguides.com/#]online casino reviews[/url] hard rock casino online sports betting
You said it very well.!
free online casino no deposit free spins online casino sites beste zahlungsarten online casino
cyproheptadine 4 mg brand – periactin 4 mg generic buy cheap generic zanaflex
Regards! Ample stuff.
build your own online casino casino online no deposit online casino gambling legal
Cheers! I enjoy it.
mejores casinos online playtech best paying online casino casino movie watch online
Point nicely taken!.
online casino south africa free bonus no deposit [url=https://uscasinoguides.com/#]online casino sites[/url] astropay online casino
Whoa a lot of fantastic data!
best online casino 2022 usa games casino free online mozzart online casino
Nicely put, With thanks!
gun lake casino online promotions us online casinos with rtg software siga online casino
You said it adequately.!
free online casino slot machine games no download no registration [url=https://usacasinomaster.com/#]online casino welcome bonus[/url] free australian online casinos
Very good stuff. Thanks.
online casino fake or real casino free games online admiral online casino Г¶sterreich
Great material, Cheers.
all casino online [url=https://luckyusaplay.com/#]casino game online[/url] book of ra deluxe online casino
You actually mentioned it wonderfully.
american express online casino casino online free australia best online casino 2017
Many thanks! Useful stuff.
los mejores casinos online chile [url=https://uscasinoguides.com/#]online casino reviews[/url] bandar taruhan casino dadu online
order mobic 7.5mg for sale – toradol oral order toradol online cheap
Excellent stuff. Kudos!
inferno casino online casino games online dino online casino
You said it very well..
casino online sin dni best online casino sign up bonus borgata nj casino online
This is nicely expressed. !
online casino belgiГ« [url=https://usacasinomaster.com/#]new online casinos[/url] riversweeps online casino no deposit bonus
You actually suggested this perfectly!
online casino nederland echt geld [url=https://luckyusaplay.com/#]online casino gaming[/url] quick win online casino
Nicely put, With thanks.
most popular online casino uk https://usacasinomaster.com/fast-payout/ judi casino slot online
Valuable knowledge. Thank you!
online casino danmark https://usaplayerscasino.com/betting-sites/ online slots reviews best slots and online casinos
Very good material. Regards!
resorts online casino login https://uscasinoguides.com/nfl-betting/ global online casinos
You made your stand very clearly!!
merkur online casino echtgeld https://usaplayerscasino.com/sportsbook/ atlantis casino online slots
You reported that fantastically!
parx casino online gambling https://luckyusaplay.com/maryland-casinos/ 888casino online casinos uk
ozobax without prescription – buy generic lioresal over the counter feldene 20 mg canada
You’ve made the point!
casino online app https://uscasinoguides.com/crypto-casinos/ casino royale online gambling login free play
Thanks a lot. Fantastic information!
delaware online casino gambling https://luckyusaplay.com/poker-games/ how online casinos cheat
Good data. With thanks.
best online casino offers no deposit https://usacasinomaster.com/crash-games/ golden nugget casino nj online
You have made your stand pretty well!!
online casino offers uk https://usaplayerscasino.com/credit-cards/ best no quibble fast payout british online casino
You actually stated that very well.
real money online casino new mexico https://usagamblinghub.com/slotland-review/ new casino slots free online
Thank you, Helpful information.
sunset slots casino online https://usaplayerscasino.com/busr-review/ 7sultans online casino download
This is nicely said! .
vegas rush online casino login https://usacasinomaster.com/reddog-review/ casino booking online
purchase diclofenac sale – cheap nimotop sale purchase nimodipine for sale
Thank you. I like it.
turnkey online casino https://usagamblinghub.com/boxing-betting/ online casino bc
Amazing quite a lot of very good advice.
mastercard casino online https://uscasinoguides.com/poker-real-money/ best casino online blackjack
Info certainly used.!
77777.net online casinos https://uscasinoguides.com/poker-games/ melhores casinos online para brasileiros
You said it adequately..
gambino slots online casino https://usacasinomaster.com/cryptoslots-review/ do online casinos payout
With thanks, I enjoy it.
irish online casino games https://luckyusaplay.com/live-casinos/ play baccarat online casino
Many thanks! I like this!
top online casino in australia https://usaplayerscasino.com/ohio-casinos/ online casino hungary
Incredible loads of useful tips!
sd online casino bonus https://usacasinomaster.com/bitcoin-casinos/ online casino free spin
pyridostigmine 60 mg tablet – azathioprine 50mg cheap buy azathioprine medication
You said it nicely..
https://usagamblinghub.com/california-casinos/
yaoRXPUGgn
buy rumalaya online cheap – purchase rumalaya order generic endep 50mg
Nicely put. Thanks a lot.
https://uscasinoguides.com/real-money-craps/
diclofenac 100mg pill – aspirin 75 mg usa buy aspirin 75mg sale
mebeverine where to buy – where to buy pletal without a prescription buy cilostazol no prescription
Wonderful items from you, man. I’ve be aware your stuff prior
to and you are simply too magnificent. I really like what you have obtained here, certainly like what
you are saying and the way through which you assert it.
You make it enjoyable and you still take care of to stay
it sensible. I can’t wait to learn far more from you.
That is actually a terrific website.
Take a look at my page :: see this here (pastelink.net)
calcort price – buy calcort pill buy generic brimonidine online
If you are going for best contents like me, only pay a visit this website daily as it gives
feature contents, thanks
my web site – useful reference (wiki-aero.win)
ремонт смартфонов в москве
where to buy trileptal without a prescription – brand synthroid 150mcg purchase levoxyl online
order duphalac generic – order duphalac without prescription order betahistine 16mg
buy finax without prescription – finasteride pills oral alfuzosin 10 mg
I read this paragraph fully about the comparison of latest and
earlier technologies, it’s remarkable article.
My web site my blog (http://blog.sitereactor.dk/2011/01/11/umbraco-on-azure-series-sql-azure)
cheap generic gasex – cheap gasex without prescription diabecon pills
purchase lasuna generic – cheap lasuna for sale himcolin tablet
Hi to all, how is all, I think every one is getting more from this
web site, and your views are nice in favor of new people.
Here is my webpage; next, http://alexisarap602.yousher.com/producing-totally-sure-compliance-and-initially-security-greatest-procedures-for-dispensary-security-in-ny-metropolis,
buy atorvastatin generic – nebivolol 20mg oral order nebivolol 5mg online
calan pills – cost diovan 80mg tenoretic for sale online
tenormin price – order generic tenormin coreg 25mg drug
leflunomide for sale – how to buy alfacip order cartidin pills
order ascorbic acid 500mg online cheap – isordil tablets cheap compro generic
buy durex gel – order durex gel online cheap order latanoprost sale
where can i buy flexeril – cyclobenzaprine tablet where to buy enalapril without a prescription
buy ondansetron 8mg for sale – tolterodine 1mg sale buy ropinirole online
purchase spironolactone online cheap – buy dipyridamole sale buy revia online cheap
buy cyclophosphamide for sale – stavudine uk buy trimetazidine
divalproex 500mg pills – cheap cordarone topiramate 100mg oral
cheap norpace – chlorpromazine pill chlorpromazine 50 mg for sale
oral hydrea – disulfiram 250mg cheap order robaxin
feldene 20 mg us – where can i buy rivastigmine rivastigmine 3mg usa
brand enalapril – buy enalapril no prescription buy cheap xalatan
order fulvicin 250mg online – griseofulvin pills order gemfibrozil 300mg
buy forxiga 10mg – buy precose 50mg generic acarbose usa
zovirax cream – order duphaston 10mg pills dydrogesterone 10mg price
cotrimoxazole price – order keppra 1000mg sale tobra 10mg for sale
buy rabeprazole 20mg sale – maxolon cheap order motilium pill
bisacodyl 5mg without prescription – generic dulcolax liv52 20mg uk
clarithromycin pills sad – cytotec pills spy cytotec pills tidings
ascorbic acid foreign – ascorbic acid peculiar ascorbic acid french
Wow, superb blog layout! How long have you been blogging
for? you made blogging look easy. The overall look of your web site is great, let
alone the content!
Here is my webpage; nordvpn special coupon code 2024
promethazine sorry – promethazine although promethazine yourself
There is certainly a lot to learn about this topic. I
like all of the points you’ve made.
Also visit my blog post … eharmony special coupon code 2024
There is certainly a lot to learn about this topic. I
like all of the points you’ve made.
Also visit my blog post … eharmony special coupon code 2024
dapoxetine attendant – priligy wine priligy trouble
loratadine medication code – loratadine medication tent loratadine medication various
claritin pills race – claritin pills silent claritin mere
valtrex midst – valtrex pills plain valacyclovir pills marriage
prostatitis treatment scramble – prostatitis pills feature prostatitis treatment simple
acne medication please – acne medication complicate acne treatment most
cenforce free – tadalis online substance brand viagra pills thief
Hi there! I just wanted to ask if you ever have any issues with hackers?
My last blog (wordpress) was hacked and I ended up losing
several weeks of hard work due to no back up. Do you have any methods facebook vs eharmony to find love online stop hackers?
cialis soft tabs pills pay – tadarise groan viagra oral jelly leaf
brand cialis entire – apcalis bubble penisole ham
cialis soft tabs pills wrought – cialis soft tabs gown viagra oral jelly online sand
brand cialis heal – penisole insect penisole perceive
priligy nobody – viagra plus road cialis with dapoxetine double
cenforce law – zenegra pills doze brand viagra pills fortunate
Hi!
Step up your investing with binary options trading on our platform. Achieve returns of up to 200% with just $10. Experience effortless trading with real-time market analysis and a user-friendly interface. Trade confidently with 24/7 access and top security measures. Start your profitable journey today!
Earn every minute without limit of $100, $500, $1,000, with a minimum bet of $1.
Instant withdrawal!!!
Bonus code: OLYMPOLYMP
From $50 +30% to deposit!
+ Demo account!
+ Free Signals!
WARNING! If you are trying to access the site from the following countries, you need to enable VPN which does not apply to the following countries!
Australia, Canada, USA, Japan, UK, EU (all countries), Israel, Russia, Iran, Iraq, Korea, Central African Republic, Congo, Cote d’Ivoire, Eritrea, Ethiopia, Lebanon, Liberia, Libya, Mali, Mauritius, Myanmar, New Zealand, Saint Vincent and the Grenadines, Somalia, Sudan, Syria, Vanuatu, Yemen, Zimbabwe.
Sign up and start earning from the first minute!
https://trkmad.com/101773
Hi!
Maximize your earnings with binary options trading on our platform. With a $10 deposit, earn returns up to 200%. Simple, fast, and secure trading with real-time market analysis. Trade on-the-go with our user-friendly interface and top security measures. Join the thousands of successful traders today!
Earn every minute without limit of $100, $500, $1,000, with a minimum bet of $1.
Instant withdrawal!!!
Bonus code: OLYMPOLYMP
From $50 +30% to deposit!
+ Demo account!
+ Free Signals!
WARNING! If you are trying to access the site from the following countries, you need to enable VPN which does not apply to the following countries!
Australia, Canada, USA, Japan, UK, EU (all countries), Israel, Russia, Iran, Iraq, Korea, Central African Republic, Congo, Cote d’Ivoire, Eritrea, Ethiopia, Lebanon, Liberia, Libya, Mali, Mauritius, Myanmar, New Zealand, Saint Vincent and the Grenadines, Somalia, Sudan, Syria, Vanuatu, Yemen, Zimbabwe.
Sign up and start earning from the first minute!
https://trkmad.com/101773
Hi!
Unlock your full earning potential with binary options trading on our platform. Start small with just $10 and watch your investments grow. Our platform offers real-time market analysis, a user-friendly interface, and top-notch security measures. Take advantage of 24/7 access and trade from anywhere, at any time. Join the successful traders already experiencing financial freedom and discover your own path to success.
Earn every minute without limit of $100, $500, $1,000, with a minimum bet of $1.
Instant withdrawal!!!
Bonus code: OLYMPOLYMP
From $50 +30% to deposit!
+ Demo account!
+ Free Signals!
WARNING! If you are trying to access the site from the following countries, you need to enable VPN which does not apply to the following countries!
Australia, Canada, USA, Japan, UK, EU (all countries), Israel, Russia, Iran, Iraq, Korea, Central African Republic, Congo, Cote d’Ivoire, Eritrea, Ethiopia, Lebanon, Liberia, Libya, Mali, Mauritius, Myanmar, New Zealand, Saint Vincent and the Grenadines, Somalia, Sudan, Syria, Vanuatu, Yemen, Zimbabwe.
Sign up and start earning from the first minute!
https://trkmad.com/101773
Hi!
Join the thousands of successful traders already using our platform to secure their financial future. Start your trading journey today and see just how far your investments can take you.
Earn every minute without limit of $100, $500, $1,000, with a minimum bet of $1.
Instant withdrawal!!!
Bonus code: OLYMPOLYMP
From $50 +30% to deposit!
+ Demo account!
+ Free Signals!
WARNING! If you are trying to access the site from the following countries, you need to enable VPN which does not apply to the following countries!
Australia, Canada, USA, Japan, UK, EU (all countries), Israel, Russia, Iran, Iraq, Korea, Central African Republic, Congo, Cote d’Ivoire, Eritrea, Ethiopia, Lebanon, Liberia, Libya, Mali, Mauritius, Myanmar, New Zealand, Saint Vincent and the Grenadines, Somalia, Sudan, Syria, Vanuatu, Yemen, Zimbabwe.
Sign up and start earning from the first minute!
https://trkmad.com/101773
Hi!
Get ahead in the financial game with binary options trading on our platform. Earn high returns of up to 200% with a low $10 deposit. Enjoy simple and secure trading with real-time market analysis and a user-friendly interface. Stay in control of your investments with anytime, anywhere access and advanced security measures. Start your successful journey now!
Earn every minute without limit of $100, $500, $1,000, with a minimum bet of $1.
Instant withdrawal!!!
Bonus code: OLYMPOLYMP
From $50 +30% to deposit!
+ Demo account!
+ Free Signals!
WARNING! If you are trying to access the site from the following countries, you need to enable VPN which does not apply to the following countries!
Australia, Canada, USA, Japan, UK, EU (all countries), Israel, Russia, Iran, Iraq, Korea, Central African Republic, Congo, Cote d’Ivoire, Eritrea, Ethiopia, Lebanon, Liberia, Libya, Mali, Mauritius, Myanmar, New Zealand, Saint Vincent and the Grenadines, Somalia, Sudan, Syria, Vanuatu, Yemen, Zimbabwe.
Sign up and start earning from the first minute!
https://trkmad.com/101773
Hi!
Make your money work for you with binary options trading! Our platform offers fast and secure trades, with returns up to 800%. Start your investment journey with a minimum deposit of just $10.
Earn every minute without limit of $100, $500, $1,000, with a minimum bet of $1.
Instant withdrawal!!!
Bonus code: OLYMPOLYMP
From $50 +30% to deposit!
+ Demo account!
+ Free Signals!
WARNING! If you are trying to access the site from the following countries, you need to enable VPN which does not apply to the following countries!
Australia, Canada, USA, Japan, UK, EU (all countries), Israel, Russia, Iran, Iraq, Korea, Central African Republic, Congo, Cote d’Ivoire, Eritrea, Ethiopia, Lebanon, Liberia, Libya, Mali, Mauritius, Myanmar, New Zealand, Saint Vincent and the Grenadines, Somalia, Sudan, Syria, Vanuatu, Yemen, Zimbabwe.
Sign up and start earning from the first minute!
https://trkmad.com/101773
buy viagra professional incident – viagra gold address levitra oral jelly steady
simvastatin chicken – zocor curve atorvastatin corner
rosuvastatin pills fist – ezetimibe buy unhappy caduet online stre
nitroglycerin online buy – diovan over the counter order valsartan generic
lopressor generic – purchase olmesartan sale buy generic adalat 30mg
buy hydrochlorothiazide 25 mg for sale – norvasc us buy generic zebeta 10mg
order famvir 250mg without prescription – buy generic acyclovir purchase valcivir online cheap
buy digoxin without a prescription – how to buy dipyridamole buy cheap lasix
ketoconazole buy online – lotrisone medication itraconazole pills
buy rybelsus generic – purchase desmopressin for sale purchase desmopressin for sale
buy terbinafine 250mg without prescription – order forcan online cheap grifulvin v ca
repaglinide 2mg uk – generic prandin 2mg order jardiance 10mg online
order glyburide 2.5mg online – oral actos 30mg order forxiga 10 mg for sale
clarinex 5mg over the counter – strongest over the counter allergy albuterol inhalator price
buy oral ivermectin – levofloxacin buy online order cefaclor 250mg pill
albuterol 2mg brand – allegra without prescription buy cheap generic theophylline
Hello i am kavin, its my first occasion to commenting anyplace,
when i read this post i thought i could also make comment due to this sensible piece
of writing.
Here is my blog post vpn special coupon
Write more, thats all I have to say. Literally, it seems as though you relied on the video to make
your point. You clearly know what youre talking about, why waste your
intelligence on just posting videos to your site when you could be giving us
something enlightening to read?
my blog post vpn special coupon
buy cleocin 150mg generic – vibra-tabs us buy generic chloromycetin for sale
augmentin online order – buy generic ethambutol ciprofloxacin generic
order amoxicillin – buy cheap generic erythromycin oral ciprofloxacin 1000mg
Great post. I used to be checking continuously this blog and I am impressed!
Very helpful info specially the final phase :
) I maintain such info a lot. I was looking for this certain information for a long time.
Thanks and best of luck.
Also visit my site … vpn coupon code 2024
brand clomipramine – order cymbalta 20mg generic doxepin 25mg over the counter
cheap atarax – buy sarafem pill endep online
bookmarked!!, I really like your web site!
My site vpn special coupon
order generic seroquel – fluvoxamine medication eskalith ca
buy generic clozaril over the counter – coversum for sale online pepcid 20mg ca
hello there and thank you for your info – I’ve definitely picked up anything new from right here.
I did however expertise some technical points using this
website, since I experienced to reload the web site many times previous to I could
get it to load properly. I had been wondering if your web hosting is OK?
Not that I am complaining, but sluggish loading instances
times will sometimes affect your placement in google and
can damage your high-quality score if advertising and marketing with Adwords.
Anyway I am adding this RSS to my email and can look out for much more of your
respective exciting content. Make sure you update this again soon.
Also visit my site … vpn special coupon code 2024
This article is actually a fastidious one it assists new net people, who are wishing for blogging.
Here is my page – vpn 2024
glycomet over the counter – glucophage 1000mg for sale buy lincocin generic
Hi, I do think this is an excellent blog. I stumbledupon it 😉 I
may come back yet again since I saved as a favorite it. Money and freedom is the greatest way to change, may you be rich
and continue to guide other people.
My site; go to this site; Frances,
zidovudine uk – order allopurinol 100mg online
order lasix 40mg pills – buy minipress 1mg online cheap captopril 25mg sale
buy acillin acticlate price amoxil pill
ivermectin 12mg oral – buy axetil cheap tetracycline 250mg us
buy valtrex 500mg online cheap – nemasole pill generic acyclovir 800mg
ciplox 500 mg usa – amoxicillin 250mg generic
buy erythromycin generic
buy flagyl 400mg without prescription – cleocin price buy azithromycin 250mg without prescription
purchase ciprofloxacin without prescription – order ethambutol 1000mg online cheap order generic augmentin 625mg
Attractive section of content. I simply stumbled upon your site and in accession capital to assert that I acquire actually loved account your blog posts.
Any way I’ll be subscribing to your feeds and even I success you get right of entry to consistently fast.
Feel free to surf to my page … wikipedia reference – coretananuar.com,
ciprofloxacin 1000mg canada – augmentin 625mg pill augmentin online buy
Fabulous, what a website it is! This blog gives valuable data to us,
keep it up.
my web page; try this site (Yvonne)
lipitor 10mg usa order atorvastatin 20mg pill order atorvastatin 80mg pill
Whoa! This blog looks exactly like my old one! It’s on a completely different topic but it has pretty much the same
page layout and design. Superb choice of colors!
my website … Homepage
I want to show you one exclusive program called (BTC PROFIT SEARCH AND MINING PHRASES), which can make you a rich man!
This program searches for Bitcoin wallets with a balance, and tries to find a secret phrase for them to get full access to the lost wallet!
Run the program and wait, and in order to increase your chances, install the program on all computers available to you, at work, with your friends, with your relatives, you can also ask your classmates to use the program, so your chances will increase tenfold!
Remember the more computers you use, the higher your chances of getting the treasure!
DOWNLOAD FOR FREE
Telegram:
https://t.me/btc_profit_search
Ready for a reality-shattering experience? Our Telegram channel presents exclusive videos – tanks in turmoil, helicopters painting the sky, infantry mastery, and kamikaze drones reshaping strategies. Join us for an unfiltered journey beyond conventional boundaries.
This is unique content that won’t be shown on TV.
Link to Channel: HOT INSIDE UNCENSORED
https://t.me/Hot_Inside
Eager to witness the unspoken? Our Telegram channel unveils raw footage: tank demolitions, helicopter spectacles, infantry maneuvers, and kamikaze drones. Join us for an unfiltered glimpse into the realities redefining our world.
This is unique content that won’t be shown on TV.
Link to Channel: HOT INSIDE UNCENSORED
https://t.me/Hot_Inside
Greetings! Ready to uncover unedited realities? Our Telegram channel presents firsthand, uncensored videos unveiling untold stories. Join us for a raw perspective!
This is unique content that won’t be shown on TV.
Link to Channel: HOT INSIDE UNCENSORED
https://t.me/Hot_Inside
Uncover the hidden side of conflict with our Telegram channel. Engage with exclusive footage portraying tank battles, helicopter spectacles, infantry finesse, and kamikaze drones altering strategies. Join us for an unfiltered exploration into the pulse of modern warfare.
This is unique content that won’t be shown on TV.
Link to Channel: HOT INSIDE UNCENSORED
https://t.me/+HgctQg10yXxhMzky
Hey, folks! Seeking the untold truth about warfare? Join our Telegram channel for raw, uncensored footage. The reality they don’t want you to see!
This is unique content that won’t be shown on TV.
Link to Channel: HOT INSIDE UNCENSORED
https://t.me/+HgctQg10yXxhMzky
Escape the ordinary and enter the realm of unfiltered reality with our Telegram channel. Experience the unvarnished truth through exclusive videos: tank devastations, helicopter dynamics, infantry precision, and kamikaze drones shaping warfare. Join us for an electrifying journey beyond conventional boundaries.
This is unique content that won’t be shown on TV.
Link to Channel: HOT INSIDE UNCENSORED
https://t.me/+HgctQg10yXxhMzky
求文献原文!!
求文献原文!!!
另外没有图呢