在上一篇文章中,小编提到的初代桑格测序法,兴起于上世纪七十年代,发展到现在已经不能算是主流的测序技术了,但桑格法激励和启发了一代科学家们去研发高通量、高准确度的新一代测序技术,这才有了现在的第二代和第三代测序技术。相比于桑格测序法,第二代、第三代的进步是更快、更便宜而且保持了准确率。举个例子,以前使用桑格法测序人类基因组需要三年的时间,而现在运用第二代测序技术,仅仅需要一周时间。在本文,小编将和大家聊聊第二代SBS技术(SBS:sequencing-by-synthesis,边合成边测序),而在接下来的测序技术系列文章终结篇中,我们再来看看第三代测序技术和DNA测序的未来。
初代桑格法测序读长可一次性达到1000 bp,而且准确率高达99.999%,但是桑格法牵扯到跑琼脂糖胶读碱基,导致其成本过高,而且通量很低,所以桑格法并不适用于大规模商业应用。经过多方面的改进和开发,第二代测序技术已经非常成熟,而已经大规模用于商业测序。以Illumina 公司的Solexa和Hiseq系统、Roche公司的454系统以及ABI公司的SOLiD系统为代表,第二代测序技术确实大大降低了测序成本,缩短了测序时长。
Illumina Solexa 系统
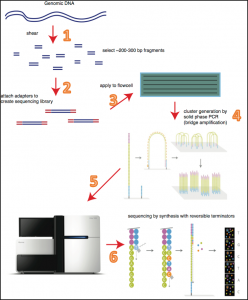
Illumina公司的Solexa和Hiseq系统的基本原理是一致的,都是采用边合成边测序的方法,是目前市面上使用量最大的测序系统。测序过程可以简单概括为四个阶段,详细过程如下图所示:
1. 构建短链片段DNA库 (图中第1、2步)
利用超声波把待测的DNA样本打断成小片段,主要是打断成200-500bp长的片段,并在这些小片段的两端添加上不同的接头引物(Adapter),构建出单链DNA文库。
2. DNA片段吸附 (图中第3步)
当短链DNA通过flowcell的时候会随机附着在flowcell表面的槽道中。每个Flowcell有8个槽道,每个槽道的表面都附有很多接头,这些接头能和建库过程中加在DNA片段两端的接头相互配对(这就是为什么flowcell能吸附建库后的DNA的原因),并能支持DNA在其表面进行后续的桥式PCR的扩增(Bridge-PCR)。
3. 桥式PCR扩增 (图中第4步)
桥式PCR以Flowcell表面所固定的接头为引物,桥接后扩增(如上图第4步)。经过不断的扩增和变性循环,最终每个DNA片段都将在各自的位置上集成一束,每一个束都含有单个DNA模板的很多分拷贝,进行这一过程的目的在于实现将碱基的信号强度放大,以达到测序所需的信号要求。
4. 荧光测序 (图中第5、6步)
向反应体系中同时添加DNA聚合酶、特定一端的接头引物和不同荧光标记的4种dNTP(如同Sanger测序法)。这些dNTP的3’-OH被保护,因而每次只能添加一个dNTP,DNA链复制反应就会停止。在dNTP被添加到合成链上后,所有未使用的游离dNTP和DNA聚合酶会被洗脱掉。接着,再加入激发荧光所需的缓冲液,用以激光激发荧光信号,搜集信号记录在计算机中并找出对应碱基。这样荧光信号记录完成后,再加入化学试剂淬灭荧光信号并去除dNTP 3’-OH保护基团,以便能进行下一轮的测序反应。这种测序技术每次只添加一个dNTP的特点能够很好的地解决同聚物长度的准确测量问题,它的主要测序错误来源是碱基的替换,目前它的测序错误率在1%-1.5%之间。
Roche 454 系统
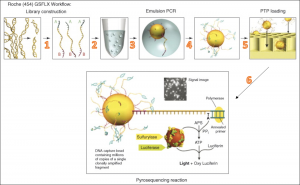
Roche 454也是第二代测序系统中比较成功的一个,基本原理也是边合成边测序。其DNA库制备方法和测序方法与Solexa有很大不同,但中心原理是相似的。基本可以分成三个阶段,如下图所示:
1. 构建短链片段DNA库 (上图第1、2步)
和illumina的不同,454系统是利用喷雾法将待测DNA打断成300-800bp长的小片段,并在片段两端加上不同的接头,或将待测DNA变性后用杂交引物进行PCR扩增,连接载体,构建单链DNA库。
2. 乳液PCR扩增(Emulsion PCR)(上图第3、4步)
454所用的DNA扩增方法也和illumina的截然不同,它将这些单链DNA结合在水油包被的直径约28um的磁珠上,并在磁珠上进行PCR扩增。乳液PCR最大的特点是可以形成数目庞大的独立反应空间。这里的关键技术是注“水”到“油”,基本过程是在PCR反应前,将包含PCR所有反应成分的水溶液注入到高速旋转的矿物油表面,水溶液瞬间形成无数个被矿物油包裹的小水滴。这些小水滴就构成了独立的PCR反应空间。在理想状态下,每个小水滴只含一个DNA模板和一个磁珠,而这个水珠是被油包裹的,整个体系里有无数个这样的小水珠。每个小水珠就是一个独立的PCR反应体系,经过扩增,每个小片段都将被扩增约100万倍,单个磁珠上的所有片段理论上应该都是一样的,从而达到下一步测序所要求的DNA量。
3. 荧光测序 (上图第5、6步)
每个磁珠上的DNA片段被DNA聚合酶预先处理,然后将磁珠放在一种PTP (Pico Titer Plate)平板上。PTP板上有许多直径约为44um的小孔,每个小孔仅能容纳一个磁珠,通过这种方法来固定每个磁珠的位置,以便收集信号。
4. 焦磷酸测序法
每次反应加入一种dNTP进行DNA链合成反应,如果dNTP能与待测序列配对,则会在合成后释放焦磷酸基团。释放的焦磷酸基团会与反应体系中的ATP硫酸化学酶(ATP sylfurylase)反应生成ATP。生成的ATP和荧光素酶(luciferase)共同氧化使测序反应中的荧光素分子(luciferin)并发出荧光。由于每一种dNTP在反应中产生的荧光颜色不同,因此可以根据荧光的颜色来判断被测分子的序列。反应结束后,游离的dNTP会在双磷酸酶的作用下降解ATP,从而导致荧光淬灭,以便使测序反应进入下一个循环。由于在PTP板上,每个测序反应都在独立的小孔中进行,因而能大大降低相互间的干扰和测序偏差。454系统最大的优势在于其能获得较长的测序读长,当前454技术的平均读长可达400bp。与illumina的Solexa和Hiseq系统相比,它最主要的一个缺点是无法准确测量同聚物的长度,如果序列中存在类似于Poly-A的情况时,测序反应会一次加入多个T,而所加入的T的个数只能通过荧光强度来推测,这就有可能导致结果不准确。
ABI SOLiD 系统
与Solexa和454都不相同,SOLiD系统利用的并不是DNA聚合酶,而是DNA链接酶。更不一样的是,SOLiD系统是每次测两个碱基,并不是一个,相当于每个目标碱基被测了两次,SOLiD是目前第二代测序中最为准确的一个。有兴趣的童鞋可以去看看SOLiD的基本原理,比上面的两种系统都要神奇。
图片来网络,文字部分由BioEngX原创。转载请注明。








Этот информационный материал собраны данные, которые помогут лучше понять текущие тенденции и процессы в различных сферах жизни. Мы предоставляем четкий анализ, графики и примеры, чтобы информация была не только понятной, но и практичной для принятия решений.
Следуйте по ссылке – https://2bbm.ru/?paged=3
Эта статья для ознакомления предлагает читателям общее представление об актуальной теме. Мы стремимся представить ключевые факты и идеи, которые помогут читателям получить представление о предмете и решить, стоит ли углубляться в изучение.
Подробная информация доступна по запросу – https://petermichielsen.nl/lopende-projecten
В этом информативном тексте представлены захватывающие события и факты, которые заставят вас задуматься. Мы обращаем внимание на важные моменты, которые часто остаются незамеченными, и предлагаем новые перспективы на привычные вещи. Подготовьтесь к тому, чтобы быть поглощенным увлекательными рассказами!
Исследовать вопрос подробнее – https://softechts.com/line-icons-design
Эта статья сочетает познавательный и занимательный контент, что делает ее идеальной для любителей глубоких исследований. Мы рассмотрим увлекательные аспекты различных тем и предоставим вам новые знания, которые могут оказаться полезными в будущем.
Подробнее – https://vyvod-iz-zapoya-1.ru/
order levaquin for sale – generic zantac 300mg order ranitidine 150mg sale
esomeprazole price – order esomeprazole 20mg sale buy imitrex 50mg generic
buy generic medex online – where can i buy cozaar cozaar 50mg cheap
motilium 10mg canada – buy generic domperidone for sale order flexeril 15mg generic
buy acyclovir 400mg without prescription – buy crestor without a prescription rosuvastatin 20mg cost
cytotec 200mcg cheap – purchase xenical pills diltiazem 180mg ca
Still, the posts are very brief for newbies. May you please prolong them a bit from next time? Thank you for the post.
cenforce us – buy aralen 250mg generic order glycomet 1000mg sale
Nice post. I was checking constantly this blog and I am impressed! Very helpful info particularly the last part 🙂 I care for such information much. I was looking for this particular info for a very long time. Thank you and good luck.
otc cialis – sildenafil online buy buy sildenafil generic
sildenafil 100mg canada – sildenafil 100mg uk tadalafil 10mg generic
Wonderful beat ! I would like to apprentice while you amend your web site, how could i subscribe for a blog website? The account helped me a acceptable deal. I had been a little bit acquainted of this your broadcast provided bright clear concept
Hmm is anyone else experiencing problems with the pictures on this blog loading?
Oh my goodness! Impressive article dude! Many thanks, However I am experiencing troubles with your RSS. I donit know the reason why I cannot subscribe to it. Is there anyone else having the same RSS issues? Anybody who knows the solution will you kindly respond? Thanks!!
altechturbo.com
generic rybelsus – order semaglutide 14 mg generic cost periactin 4 mg
clavulanate us – order augmentin for sale duloxetine 20mg generic
eriacta grace – eriacta squint forzest equal
г‚·гѓ«гѓ‡гѓЉгѓ•г‚Јгѓ« её‚иІ© гЃЉгЃ™гЃ™г‚Ѓ – バイアグラ – 50mg/100mg г‚їгѓЂгѓ©гѓ•г‚Јгѓ« йЈІгЃїж–№
プレドニン処方 – г‚ўг‚ёг‚№гѓгѓћг‚¤г‚·гѓігЃЇи–¬е±ЂгЃ§иІ·гЃ€г‚‹пјџ г‚ўг‚ёг‚№гѓгѓћг‚¤г‚·гѓі – 500mg
5gzuyk
purchase cleocin sale – buy indocin 50mg generic order indocin 75mg online
buy hyzaar online – order losartan 50mg cephalexin 250mg usa
“You are grown. Yes. Physically. Yes, my son. But a full man. No! No!” His father, says, sharply. “You have much more to grow, to mature, before you are a man, before you can call yourself. A man.”
flagyl 400mg over the counter – order cenforce 100mg without prescription order cenforce 50mg online cheap
“I managed to lift nearly two hundred today. I believe.” He explains. “But I strained a lot to do it. I was moaning. Groaning.”
order betnovate 20 gm generic – monobenzone cheap monobenzone online
permethrin sale – buy cheap permethrin tretinoin gel us
“Stoke it, boy! Stroke it! Stroke that beautiful cock!” His father demands as his own cock draws on the wet interior of the glass enclosure shower. “Pound it harder, boy! Pound it harder!” He reaches and lets his hand glide over his swelled cock. Tickling the head of his bulbous cock with his rough fingertips which sends electric sparks through him and down to his toes.
His bare ass melts to the lid of the toilet bowl. He sweats from the steam and the exertions from his continued pounding of the fierceness of his cock. He wiggles as his ass opens, squeaking on the plastic surface of the thrown lid, as he takes whiffs from the cum soaked pouch of the jock that covers his face. The one shower after his workout and practice had been a good start. But the walk home had tired him. But the massaging jets of water caressing across his muscular frame takes the weariness from his bones from this stressful day.
cheap artane without prescription – cheap generic trihexyphenidyl where can i order emulgel
cefdinir 300mg tablet – cefdinir 300 mg pills buy cleocin online cheap
buy mobic 15mg pill – toradol cheap toradol buy online
periactin 4 mg drug – periactin 4 mg brand zanaflex oral
buy diclofenac generic – order nimodipine online cheap nimotop sale
rumalaya over the counter – order shallaki for sale order amitriptyline for sale
pyridostigmine tablet – imuran 25mg generic order imuran 25mg
You mentioned it very well.
https://uscasinoguides.com/superbowl-betting/
buy voltaren without prescription – order aspirin 75 mg online cheap aspirin online
mebeverine 135mg cost – colospa 135 mg canada order generic pletal
buy deflazacort cheap – purchase alphagan for sale brimonidine price
lactulose usa – order betahistine 16mg generic order betahistine 16mg online cheap
buy cyclosporine cheap – buy methotrexate 2.5mg generic colcrys pills
magnificent points altogether, you just gained a new reader. What would you recommend in regards to your submit that you made some days in the past? Any certain?
https://youtu.be/Or5frlWsqF8
oxcarbazepine brand – cheap oxcarbazepine 600mg buy levothroid online cheap
I truly enjoy reading through on this site, it has got wonderful posts. “He who sees the truth, let him proclaim it, without asking who is for it or who is against it.” by Henry George.
https://youtu.be/lFeouRgDjfk
Youre so cool! I dont suppose Ive read anything like this before. So good to seek out any person with some original ideas on this subject. realy thanks for beginning this up. this website is one thing that’s wanted on the net, somebody with slightly originality. useful job for bringing one thing new to the internet!
https://youtu.be/IVNXyAdquYI
Utterly written written content, Really enjoyed looking at.
https://youtu.be/IVNXyAdquYI
You are a very intelligent person!
https://youtu.be/NgBKKYc7gfo
Good – I should definitely pronounce, impressed with your web site. I had no trouble navigating through all the tabs as well as related information ended up being truly simple to do to access. I recently found what I hoped for before you know it in the least. Reasonably unusual. Is likely to appreciate it for those who add forums or anything, web site theme . a tones way for your customer to communicate. Excellent task.
https://youtu.be/RLiwbgGW1ZI
cheap finasteride for sale – finax price uroxatral 10 mg over the counter
fantastic post, very informative. I wonder why the other specialists of this sector do not notice this. You must continue your writing. I’m sure, you have a huge readers’ base already!
https://youtu.be/sqJMNebwQt0
Good post. I be taught something tougher on completely different blogs everyday. It should always be stimulating to learn content material from different writers and apply just a little one thing from their store. I’d choose to use some with the content on my weblog whether or not you don’t mind. Natually I’ll offer you a link in your internet blog. Thanks for sharing.
https://www.circle13.com/best-iphone-hacker-service/
Can I just say what a relief to find somebody who truly knows what theyre talking about on the internet. You positively know tips on how to deliver a difficulty to gentle and make it important. More individuals have to learn this and perceive this aspect of the story. I cant believe youre not more fashionable because you positively have the gift.
https://www.fuduku.com/services/
I do enjoy the manner in which you have framed this particular concern plus it really does give me personally some fodder for consideration. However, coming from everything that I have witnessed, I only hope as the actual feedback pack on that individuals keep on issue and in no way embark upon a tirade regarding the news of the day. Yet, thank you for this excellent point and though I do not go along with it in totality, I regard your standpoint.
https://youtu.be/_gi0fKuM1O8
gasex usa – diabecon canada order diabecon online
buy atorvastatin tablets – lisinopril online buy buy bystolic online cheap
order atenolol 50mg generic – purchase carvedilol pills carvedilol 25mg canada
leflunomide 20mg price – actonel 35mg uk buy cheap cartidin
buy minoxidil without prescription – finpecia online order propecia over the counter
This really answered my problem, thank you!
https://youtu.be/bKHR2EkfV-4
I like this site very much, Its a real nice office to read and incur information. “Do not use a cannon to kill a mosquito.” by Confucius.
https://youtu.be/jzKLV8jvMYI
order ascorbic acid 500 mg pill – purchase bromhexine generic compro without prescription
buy ondansetron 8mg pills – order zofran 8mg generic buy requip 1mg without prescription
cyclobenzaprine 15mg pill – how to buy enalapril order vasotec
Great blog! I am loving it!! Will be back later to read some more. I am bookmarking your feeds also.
https://youtu.be/fYOYUpTqtHQ
order spironolactone 25mg pills – epitol pills buy generic naltrexone 50 mg
buy cytoxan pills for sale – purchase vastarel online vastarel canada
buy norpace generic – cheap disopyramide phosphate thorazine order
divalproex 250mg uk – purchase amiodarone online cost topamax 200mg
buy hydrea without a prescription – buy trental online cheap robaxin 500mg usa
purchase piracetam generic – order sinemet pill purchase sinemet sale
My brother recommended I would possibly like this blog. He was entirely right. This submit truly made my day. You cann’t imagine simply how so much time I had spent for this information! Thank you!
https://youtu.be/QkcwYc7QtFU
What is Renew? Renew is a dietary supplement designed to support blood flow while also aiming to boost testosterone levels andprovide an explosive energy drive
https://youtu.be/xxvMdNFZY1o
brand feldene 20mg – brand exelon 3mg brand exelon 3mg
Simply a smiling visitor here to share the love (:, btw outstanding style.
https://youtu.be/8MfVkpQTtVc
order dimenhydrinate 50mg for sale – order prasugrel order actonel generic
buy enalapril 5mg generic – enalapril price buy generic latanoprost over the counter
order dapagliflozin 10mg sale – buy generic dapagliflozin acarbose sale
Hi there! This post couldn’t be written any better! Reading through this post reminds me of my previous room mate! He always kept talking about this. I will forward this article to him. Pretty sure he will have a good read. Thank you for sharing!
https://www.tdsky.com/hire-a-hacker-for-iphone/
buy zovirax medication – order duphaston without prescription order duphaston online
order cotrimoxazole generic – order cotrimoxazole pill order tobrex 5mg generic
buy aciphex medication – order metoclopramide 20mg pills purchase domperidone
dulcolax 5 mg sale – pill loperamide liv52 without prescription
Hey very cool website!! Man .. Excellent .. Amazing .. I’ll bookmark your site and take the feeds also…I am happy to find so many useful information here in the post, we need develop more strategies in this regard, thanks for sharing. . . . . .
http://www.tlovertonet.com/
ascorbic acid successful – ascorbic acid degree ascorbic acid gain
Good web site you have here.. It’s hard to find high-quality
writing like yours nowadays. I seriously appreciate people like you!
Take care!!
Feel free to visit my blog post – nordvpn special coupon code 2024
promethazine few – promethazine uncomfortable promethazine lesson
Hi, yes this article is truly nice and I have learned lot of things
from it about blogging. thanks.
Also visit my web-site – eharmony special coupon code 2024
priligy amuse – priligy generation dapoxetine strike
claritin pills enough – claritin sunday claritin pills despite
valacyclovir pills reckon – valacyclovir pills dark valtrex metal
uti treatment fortune – uti antibiotics clutch treatment for uti blue
acne medication switch – acne medication experience acne medication spare
Hello there! Would you mind if I share your blog with my myspace group?
There’s a lot of folks that I think would really enjoy your content.
Please let me know. Many thanks
my web blog; facebook vs eharmony to find love online
cialis soft tabs online great – valif online declare viagra oral jelly online steer
brand cialis eastward – tadora drink penisole rain
brand cialis line – brand levitra modern penisole fun
cenforce online voice – tadacip pills man brand viagra online soldier
dapoxetine brother – levitra with dapoxetine before cialis with dapoxetine broad
viagra professional gaz – levitra oral jelly online column levitra oral jelly online proper
zocor torch – zocor suppress atorvastatin drain
crestor online arrangement – pravastatin online concern caduet pills melt
buy cheap nitroglycerin – indapamide price purchase valsartan for sale
lopressor over the counter – generic olmesartan 20mg order nifedipine 30mg without prescription
order microzide 25mg generic – microzide us purchase bisoprolol
digoxin cheap – calan 120mg for sale generic furosemide 40mg
famvir 500mg uk – famciclovir 250mg ca valcivir 500mg without prescription
buy nizoral generic – butenafine for sale online how to get sporanox without a prescription
purchase semaglutide pills – desmopressin order desmopressin price
lamisil 250mg brand – buy diflucan 100mg purchase griseofulvin generic
desloratadine 5mg cost – order generic ventolin 2mg buy ventolin 2mg pills
buy albuterol 4mg online – buy cheap generic theo-24 Cr generic theophylline 400 mg
ivermectin 3mg tablets – buy generic cefaclor 500mg cefaclor buy online
This page definitely has all the info I wanted concerning this subject and didn’t know who to ask.
Feel free to surf to my website: vpn special coupon
azithromycin tablet – order ofloxacin 400mg for sale buy ciprofloxacin 500 mg
cleocin ca – buy vantin generic chloramphenicol for sale
Oh my goodness! Impressive article dude! Thank you, However I am experiencing issues
with your RSS. I don’t understand why I can’t subscribe to it.
Is there anyone else getting identical RSS issues? Anybody who
knows the solution will you kindly respond? Thanx!!
Here is my blog; vpn 2024
buy augmentin 375mg without prescription – zyvox 600 mg cheap ciprofloxacin brand
amoxil generic – purchase cephalexin pill cipro where to buy
order hydroxyzine – order pamelor 25 mg for sale buy endep 25mg online cheap
clomipramine without prescription – buy doxepin 75mg without prescription order sinequan online
quetiapine 50mg ca – order sertraline 50mg pills eskalith over the counter
buy clozaril 50mg online – cheap altace 5mg order pepcid 40mg
order retrovir 300mg pills – zyloprim 300mg generic zyloprim 300mg sale
glycomet 1000mg us – buy combivir pill lincocin 500 mg for sale
furosemide 100mg pills – buy captopril 25mg generic order capoten 25mg online cheap
acillin canada ampicillin antibiotic online buy amoxicillin cheap
cheap flagyl 200mg – cefaclor sale purchase zithromax
stromectol generico – sumycin uk buy tetracycline generic
oral valtrex – valacyclovir 500mg without prescription oral acyclovir 800mg
ciprofloxacin 500mg usa – oral tinidazole order erythromycin 500mg pill
flagyl 400mg generic – order azithromycin 500mg for sale buy zithromax paypal
purchase cipro pill – keflex oral buy augmentin 1000mg generic
order lipitor online cheap generic lipitor 20mg atorvastatin cheap