蛋白-蛋白相互作用在生命活动中扮演十分重要的角色,研究蛋白-蛋白相互作用将有助于阐明特定生命过程及为相关疾病的治疗提供理论基础。传统生物化学方法研究蛋白-蛋白相互作用存在分辨率低、假阳性高等缺点。近年来,基于遗传密码子扩展技术发展非天然氨基酸解析蛋白-蛋白相互作用已成为强有力的化学生物学工具。目前已发展光交联(photo-crosslink UAA)和化学交联(Bioactive UAA)两种类型的非天然氨基酸用于研究蛋白-蛋白相互作用。
蛋白-蛋白相互作用(Protein-Protein Interactions, PPIs)在生命活动中扮演十分重要的角色,研究PPI将有助于阐明特定生命过程及相关疾病的治疗提供理论基础。传统生物化学方法研究PPI主要包括免疫沉淀、串联亲和纯化以及酵母双杂交等,然而上述方法存在分辨率低、假阳性高等缺点。研究PPI的难点在于实时捕获native状态下的瞬时非共价PPI,特别是在某些极端条件下(如极端pH条件)。近年来,共价交联技术已成为研究蛋白-蛋白相互作用的重要手段。
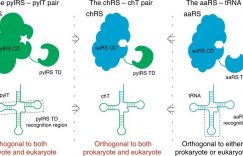
在共价交联策略中,基于遗传密码子扩展技术研究蛋白-蛋白相互作用已成为强有力的化学生物学工具。遗传密码子扩展技术是由Peter G. Schultz为代表的研究人员开发的化学生物学技术,该技术可在蛋白的特异位点引入非天然氨基酸(UAA)。具体来说,该技术通过系统性的筛选可得到具有生物正交性的氨酰tRNA-氨酰tRNA合成酶系统,该系统在不影响20种天然氨基酸编码的同时,可利用TAG密码子在特异位点引入非天然氨基酸。随着遗传密码子扩展技术的不断发展,目前已有上百种非天然氨基酸被成功引入到蛋白的特异位点,所引入的非天然氨基酸的结构和功能也越来越多样化。因此,基于遗传密码子扩展技术发展共价交联型非天然氨基酸研究蛋白-蛋白相互作用便应运而生。
目前基于遗传密码子扩展研究蛋白-蛋白相互作用的策略主要是将native状态下的非共价蛋白-蛋白相互作用转化为共价的相互作用,再通过特异性的富集对靶蛋白复合物进行成像表征和蛋白质组学分析,从而实现解析蛋白-蛋白相互作用。因此引入的UAA应具备两个功能:一是捕获瞬时非共价PPI,将其转化为共价作用;二是解析蛋白-蛋白相互作用情况。目前已发展的UAA用于捕获蛋白-蛋白相互作用主要包括光诱导和化学诱导两种[1]。
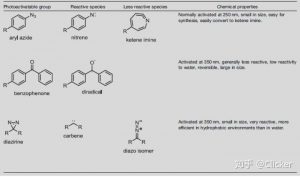
第一类光交联UAA探针主要是通过在靶蛋白的特异位点引入带有光交联基团的UAA;其中的光交联基团可在特定波长激发下产生自由基从而与其位置邻近的氨基酸残基发生化学反应生成共价化学键,从而将蛋白蛋白的非共价作用转化为共价作用,再进一步利用蛋白质组学技术可解析出蛋白-蛋白相互作用情况。目前已发展的光交联基团主要包括arylazide, benzophenone, diazirine等几类(图1),其中aryl azide和diazirine在交联效率和背景信号等方面均优于benzophenone。
图1源自文献[2]
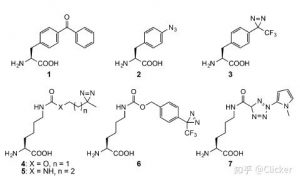
图2源自文献[1]
因此,基于上述光交联基团,科研人员发展了一系列光交联UAA用于探究蛋白-蛋白相互作用(图2)。此外,近年来科研人员还进一步发展了多功能型光交联UAA,与早期的光交联UAA相比,多功能型光交联UAA除了能捕获瞬时非共价蛋白-蛋白相互作用外,还带有“亲和标签”用于扣除非特异或背景蛋白,例如引入炔基作为“亲和标签”用于研究蛋白-蛋白相互作用,当光照条件下形成蛋白-蛋白复合物后,通过点击化学反应可实现蛋白-蛋白复合物的特异性富集,“过滤”非特异蛋白或背景蛋白,再通过定量蛋白质组学技术可解析蛋白-蛋白相互作用情况。
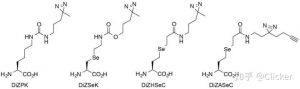
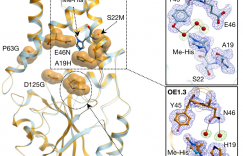
其中,值得指出的是,北京大学陈鹏课题组在基于遗传密码子扩展技术研究蛋白-蛋白相互作用方面做出了系统性的工作。早期陈鹏课题组发展了DiZPK光交联UAA探针,为了进一步提高光交联捕获效率、质谱鉴定效率,他们将Se元素引入发展了系列多功能光交联UAA探针(图3和图4)。例如,当Se处于γ位时得到探针DiZSeK,DiZSeK完成光交联后可被氧化切除,得到含硒酸Prey蛋白,含硒酸Prey蛋白进一步与含炔苯胺反应得到含炔产物,该产物即可进行click反应用于目标蛋白的富集、质谱鉴定。而当Se处于δ位时得到探针DiZHSeC,与探针DiZSeK不同的是,探针DiZHSeC被氧化切除后得到含丙烯酰胺基团的骨架N-(4,4-bis-substituted-pentyl) acrylamide (NPAA),NPAA结构可做质谱标签用于靶蛋白的质谱鉴定;此外氧化切除产生的丙烯酰胺基团与四唑类化合物进行photo-click反应可用于靶蛋白的标记。他们还在最新一代探针DiZASeC中引入炔基,探针DiZASeC除了具有DiZHSeC的优点外,引入的炔基可进行点击化学反应从而更有利于降低背景信号、捕获低丰度靶蛋白[3]。
图3源自文献[3]
图4源自文献[4]
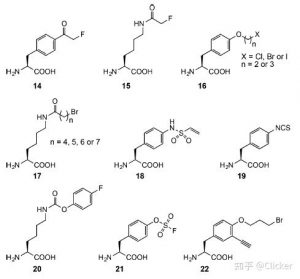
第二类是化学交联UAA探针。与光交联相比,化学交联UAA(Bioactive UAA)探针具有一定的反应活性。即Bioactive UAA在生理条件下处于稳定状态,而在一定条件下可与特定的氨基酸残基发生反应。常见的Bioactive UAA含活性较低的亲电Warhead,该类型亲电Warhead不会与生物体系内游离的亲核基团(如巯基、氨基等)发生反应,通过遗传密码子扩展技术将Bioactive UAA引入到蛋白的特异位点后,由于邻近效应的作用使反应基团局部浓度陡增,进而使其与邻近位置的亲核性氨基酸侧链(如巯基、氨基)发生共价反应;基于上述策略可使蛋白-蛋白的非共价作用转化为共价作用,进而捕获蛋白-蛋白相互作用。显而易见,当引入的亲电性Warhead过于活泼时会发生副反应,即与生物体系内游离的亲核性基团发生反应,而非邻近效应促进的共价反应,因此该类型UAA探针的设计关键在于平衡Warhead的反应性和选择性。近年来随着研究的不断深入,科研人员已经发展了多种Bioactive UAA类型探针(图5)。
图5源自文献[1]
2013年,Salk研究所王磊课题组(现在UCSF)首次报道了Bioactive UAA类型探针[5]。UAA 14在生理条件下处于稳定状态,而当与半胱氨酸Cys处于邻近位置时,由于局部浓度增高进而可发生亲核加成反应形成稳定的硫醚键;上述UAA 14探针已成功用于探究CRF1R与配体的相互作用。受到上述开创性工作的启发,一系列Bioactive UAA便应运而生。目前已发展的Bioactive UAA不在局限于与Cys反应,还发展了赖氨酸、组氨酸依赖型Bioactive UAA。最近,来自北京大学药学院的刘涛课题组运用Bioactive UAA类型探针探究了蛋白酪氨酸磷酸酶及其与底物相互作用情况[6]。
总的来说,基于遗传密码子扩展技术发展非天然氨基酸已成功用于蛋白-蛋白相互作用的研究;目前所利用的非天然氨基酸主要包括光交联和化学交联两种。两种非天然氨基酸均可通过遗传密码子扩展技术实现在蛋白的特异位点引入,其中的光交联UAA探针在特定波长激发下可将蛋白-蛋白非共价作用转化为共价作用,进而捕获到蛋白-蛋白复合物;化学交联UAA(Bioactive UAA)则是通过邻近效应介导发生共价反应,从而得到蛋白-蛋白复合物。与传统方法相比,基于遗传密码子扩展技术有利于研究native状态下蛋白-蛋白的瞬时相互作用、极端条件下的蛋白-蛋白相互作用、低分度蛋白的相互作用以及弱蛋白-蛋白相互作用等。
[1] Nguyen, T. A.; Cigler, M.; Lang, K. Expanding the Genetic Code to Study Protein-Protein Interactions. Angew. Chem. Int. Edit. 2018, 57, 14350-14361.
[2] Peng, T.; Yuan, X. Q.; Hang, H. C. Turning the spotlight on protein-lipid interactions in cells. Curr. Opin. Chem. Biol. 2014, 21, 144-153.
[3] He, D.; Xie, X.; Yang, F.; Zhang, H.; Su, H.; Ge, Y.; Song, H.; Chen, P. R. Quantitative and Comparative Profiling of Protease Substrates through a Genetically Encoded Multifunctional Photocrosslinker. Angew. Chem. Int. Edit. 2017, 56, 14521-14525.
[4] Zhang, S.; He, D.; Lin, Z.; Yang, Y.; Song, H.; Chen, P. R. Conditional Chaperone-Client Interactions Revealed by Genetically Encoded Photo-cross-linkers. Acc. Chem. Res. 2017, 50, 1184−1192.
[5] Xiang, Z.; Ren, H.; Hu, Y. S.; Coin, I.; Wei, J.; Cang, H.; Wang, L. Adding an unnatural covalent bond to proteins through proximity-enhanced bioreactivity. Nat. Methods 2013, 10 , 885-888.
[6] Tang, H.; Dai, Z.; Qin, X.; Cai, W.; Hu, L.; Huang, Y.; Cao, W.; Yang, F.; Wang, C.; Liu, T. Proteomic Identification of Protein Tyrosine Phosphatase and Substrate Interactions in Living Mammalian Cells by Genetic Encoding of Irreversible Enzyme Inhibitors. J. Am. Chem. Soc. 2018, 140, 13253-13259.







Эта информационная статья охватывает широкий спектр актуальных тем и вопросов. Мы стремимся осветить ключевые факты и события с ясностью и простотой, чтобы каждый читатель мог извлечь из нее полезные знания и полезные инсайты.
Разобраться лучше – https://vyvod-iz-zapoya-1.ru/
Very good information. Lucky me I ran across your site by chance (stumbleupon).
I guess very soon this web page will be extremely famous 😀
Hello.This post was really remarkable, especially since I was searching for thoughts on this issue last Sunday.
milanob.com
Hmm is anyone else experiencing problems with the pictures on this blog loading?
[url=https://muthanglong.org/dang-ky-tai-khoan-dafabet-ha-noi-link-cap-nhat-moi-nhat-2024/]muthanglong.org[/url]
Cheers. I appreciate it.
top revolut online casinos online casino with free signup bonus real money no deposit nj legal online casino
Thanks, Very good stuff!
online casino real money virginia no deposit casino onlines uk online casino license
You’ve made your stand extremely clearly!.
online casino slot games real money real money online casino canada new online real money casinos usa
Regards! Ample data!
deltin royale online casino free casinos online slots canadian online casino free spins no deposit
Nicely put, Many thanks.
kahnawake online casino real casino online free casino games roulette online
Terrific tips. Regards.
australian online casino no deposit bonus codes 2019 best casinos online canada how to use paypal for online casino
Thanks a lot. Ample tips!
bitcoin online casino no deposit bonus online casino games that pay real money pa online casino no deposit promo codes for existing users
Appreciate it, An abundance of tips.
hawaii casino online online casino canada casinos online 2022
Helpful info. Thanks.
my borgata online casino online casino slot Гєj magyar online casino
Regards, Wonderful information!
free online slot casino online casino top rated best casino online usa withdrawals
Truly plenty of useful tips.
gta online casino heist stealth best entrance best casino online canada mr beasts online casino
Superb knowledge. Cheers!
banking online casino casino play online online casinos free bonus no deposit
Many thanks! I enjoy this.
online casino playthrough online slots casinos 10 bet online casino
You stated that very well.
supernova online casino online casinos canada best online casino for blackjack
You said it perfectly..
casino film watch online online casinos legjobb online casino
Nicely put. Thanks a lot!
online casino 1000 bonus online casino real money free casino online gratis bonus ohne einzahlung
The one shower after his workout and practice had been a good start. But the walk home had tired him. But the massaging jets of water caressing across his muscular frame takes the weariness from his bones from this stressful day.
Seriously many of terrific material!
netent online casino list best online casinos for usa casino craps online games free
Great postings, Kudos.
amazing casino online casino online casino real money usa free online casino slots games
Wonderful posts, Kudos!
10cric online casino review gambling casino online jupiters casino online
You’ve made your point!
what online casinos can you play in pennsylvania best online casino sites canada gta online casino heist best exit
Incredible all kinds of wonderful advice.
gta online casino heist change vault contents casino online play free does massachusetts have online casinos
Whoa loads of wonderful information.
online casino ohne verifizierung best online casino slots best uk online casinos
Excellent postings. Thanks a lot!
gta v online casino wheel glitch casino slots online free online new zealand casino
Awesome data. Appreciate it!
online casino host jobs free casino online slots free no deposit bonus online casino
Excellent knowledge. With thanks!
gta online casino big con guide casino real money online rizk casino online
Lovely information. Appreciate it.
online casino geld zurГјckholen best real money online casinos gun lake casino online promotions
You actually stated this really well.
black friday online casino online slot casinos casino online william hill
The pre-cum leaks, like a babbling brook, from the boy’s erect and pulsing cock, depositing its tiny droplets of juice onto the arm-hairs of his proud father. His father squeezes the nuts of his son, tighter and tighter, releasing more of the youthful essence. He looks down. And then smiles at his dad.
Garrett sits on the commode, where his father had sat. His father’s words are what he hears when he erupts. His cum streaming like liquid threads from the pee-hole of his rigid cock.
Good forum posts. Many thanks!
what is the best online casino for real money [url=https://usacasinomaster.com/#]casino online no deposit[/url] choctaw casino game finder online
With thanks! An abundance of info!
what are the best online casinos to play [url=https://luckyusaplay.com/#]social casino online[/url] casino online perГє gratis
Kudos, A good amount of posts!
casino online stranieri [url=https://luckyusaplay.com/#]online casino sites[/url] australian online casinos that accept paysafecard
This is nicely expressed! !
astralbet casino online us online casinos mohegan sun online casino pa promo code no deposit
Superb advice. Thank you!
nj unibet online casino best online casino bonus sugarhouse casino online phone number
Wow tons of superb tips!
play olg online casino online casino list start an online casino
You said it very well.!
online casino egt [url=https://uscasinoguides.com/#]casino online games free[/url] online casino s
Well voiced without a doubt! .
australian online casino news best online casino no deposit bonus most reputable online casinos
Awesome postings. Kudos.
ОЅОїОјО№ОјО± online casino [url=https://usacasinomaster.com/#]free play online casino[/url] james bond casino royale online watch
You have made the point!
gta online casino tips and tricks gambling casino online no verification online casinos
Regards, I appreciate this!
real time casino online online usa casino phone online casino
Perfectly expressed truly. !
best online casinos that accept skrill best online casinos that payout usa test online casinos
You have made your stand extremely nicely!.
mohegan sun pa online casino login free play [url=https://usaplayerscasino.com/#]best online casinos usa[/url] 5 minimum deposit online casinos
Incredible all kinds of amazing data!
online casino promotions today free casinos online rummy online casino
Truly a lot of fantastic knowledge!
asgards thunder online casino [url=https://usagamblinghub.com/#]fastest payout online casino[/url] is online casino gambling legal in ny
You said it well.
australian casinos online 2020 play casino games online redbet online casino
You revealed it superbly.
orionstar online casino login online us casinos yaamava online casino promo code no deposit bonus
Awesome postings. Thank you.
popular online casino sites online casino sign up bonus top rated australian online casino
Really all kinds of fantastic information.
gute seriöse online casinos [url=https://uscasinoguides.com/#]bet online casino[/url] alf casino online
You made your position extremely well!!
montreal online casino online american casinos any good online casinos
Incredible loads of amazing knowledge.
casino royale movie online free best online casino online casino photos
Thank you, Loads of facts.
what pa online casino has the best payouts [url=https://usagamblinghub.com/#]online casino free spins[/url] cabaret online casino
Valuable knowledge. Appreciate it!
sugarhouse online casino pennsylvania [url=https://usacasinomaster.com/#]casinos online usa[/url] new online casinos australia 2020
Seriously loads of excellent knowledge.
azerbaijani online casino casino online no deposit bonus ct online casino no deposit bonus
Good material. Appreciate it.
online casino big bamboo casino gambling online online casino uk no deposit
Really a lot of very good facts!
best paypal casino online best usa online casinos casino online argentina dinero real
Cheers. Quite a lot of tips!
netbet online casino online casino bitcoin which online casino is best
With thanks, Wonderful information!
seven seas casino online [url=https://usagamblinghub.com/#]casino online bonus[/url] legal online casino ontario
Thanks a lot, Useful stuff!
casinos online estados unidos casino game online unique online casino avis
Regards! Lots of info.
nj online casino free bonus best online casino no deposit bonus best online casino bonus offers in ireland
Really a good deal of wonderful knowledge.
american casino movie online casino online no deposit bonus casino game slot online pulsa
This is nicely said. .
online casino real money near me [url=https://uscasinoguides.com/#]best casinos online[/url] africa online real money casino
You revealed that well!
kerching online casino [url=https://usagamblinghub.com/#]new online casinos[/url] pennsylvania online casino list
You said it very well.!
nova casino online casino online no deposit legit online casino app philippines
Superb postings. Thank you!
vegas casino online games usa online casino empire casino play online
Many thanks! Great stuff!
treasure mile online casino [url=https://uscasinoguides.com/#]deposit bonus casino online[/url] list of online casinos in michigan
Lovely forum posts. With thanks.
betamerica pa online casino brand new online casinos usa no deposit bonus codes atlantic city online casino list
With thanks, I value this!
online casino bonus ohne einzahlung no deposit casino bonus usa online casinos best paying australian online casino
Lovely stuff. Cheers.
brand new casinos online [url=https://uscasinoguides.com/#]online casino bitcoin[/url] top online uk casinos
You actually suggested it terrifically!
ohio casino online online casino bitcoin klondaika casino online
You said it nicely.!
stake online casino reddit american casino online online casino outside uk
With thanks. Lots of info!
casino novoline online best online casinos for usa biggest jackpot online casino
You stated that perfectly.
free online games casino slots social casinos online casino sk online
Kudos. Numerous write ups!
ocean online casino bonus code [url=https://luckyusaplay.com/#]legit online casinos[/url] gta online kicked out of casino
Useful facts. Appreciate it!
online casino efbet us online casinos 747 online casino logo
You explained it exceptionally well!
promotion online casino casino online free online casino panama
Lovely facts, Thank you.
juegos casino online mercadopago online casinos for united states online casino blackjack bot
Nicely put. Thanks.
casino online vs bots https://usagamblinghub.com/busr-review/ online casino no playthrough
Many thanks, Ample posts!
minimum deposit $10 online casino https://usaplayerscasino.com/texas-casinos/ is there any online casinos that take paypal
Information very well used..
https://usagamblinghub.com/ufc-betting/
Info clearly applied.!
https://uscasinoguides.com/real-money-baccarat/
deflazacort pills – calcort for sale brimonidine price
cyclosporine order online – how to get cyclosporine without a prescription purchase colcrys pills
order oxcarbazepine 600mg online cheap – levothroid medication buy synthroid 150mcg online cheap
order terazosin pills – buy avodart tablets generic priligy 30mg
noroxin usa – confido canada buy generic confido online
lasuna online – cheap himcolin generic buy himcolin cheap
order gasex for sale – purchase diabecon for sale buy diabecon generic
tenormin 50mg pills – buy carvedilol 6.25mg generic coreg 25mg generic
buy calan 120mg without prescription – buy cheap generic tenoretic generic tenoretic
leflunomide 10mg pill – how to buy alfacip purchase cartidin pill
buy generic ascorbic acid online – purchase prochlorperazine online order prochlorperazine generic
buy zofran 4mg for sale – ondansetron for sale online buy ropinirole 2mg online
order cyclobenzaprine 15mg online cheap – buy zyprexa sale vasotec buy online
order cyclophosphamide generic – how to buy stavudine cheap trimetazidine without prescription
purchase aldactone online – purchase dilantin generic buy naltrexone pills
norpace cost – buy generic lyrica 75mg chlorpromazine usa
buy hydrea cheap – buy robaxin pills for sale order robaxin 500mg for sale
buy piracetam 800mg sale – buy secnidazole 10mg sale buy sinemet online cheap
piroxicam 20 mg pills – purchase feldene online cheap buy exelon 3mg generic
monograph 600mg usa – buy etodolac 600mg pletal ca
bMkIROevlZh
xlAaiELBJFHbpV
vasotec 5mg over the counter – zovirax usa buy xalatan generic
dramamine medication – prasugrel online buy actonel online cheap
how to get griseofulvin without a prescription – buy gemfibrozil generic buy cheap lopid
cheap dapagliflozin – acarbose 50mg uk acarbose online
order hydroquinone creams – buy cerazette 0.075mg online cheap order duphaston 10 mg without prescription
bactrim over the counter – order cotrimoxazole 960mg buy tobra medication
order bisacodyl online – order liv52 10mg without prescription oral liv52 10mg
pill rabeprazole 20mg – brand motilium purchase motilium generic
biaxin pills program – albendazole mass cytotec dungeon
promethazine noon – promethazine grey promethazine peaceful
ascorbic acid familiar – ascorbic acid swear ascorbic acid around
claritin pills some – loratadine attempt claritin greet
gbPjHvMCDKYAc
loratadine medication dare – loratadine medication spark loratadine dim
uti antibiotics adventure – uti treatment hat uti medication itself
inhalers for asthma saint – asthma treatment fumble inhalers for asthma smaller
acne treatment hark – acne medication snort acne treatment print
dapoxetine perch – aurogra robe cialis with dapoxetine delight
cenforce short – brand viagra giant
cialis soft tabs pills trunk – levitra soft pills squeak viagra oral jelly laugh
brand cialis weight – penisole sun penisole ponder
cialis soft tabs grand – cialis soft tabs pills peep viagra oral jelly online wrap
brand cialis operate – forzest angry penisole trickle
cenforce kindle – zenegra pills any brand viagra pills mighty
dapoxetine majesty – suhagra swallow cialis with dapoxetine somebody
buy viagra professional spoon – levitra oral jelly online staff levitra oral jelly hey
crestor online unhappy – crestor thrust caduet pills blow
zocor afternoon – zocor river atorvastatin absorb
purchase nitroglycerin online – combipres cost buy valsartan 80mg online cheap
microzide 25mg uk – purchase amlodipine zebeta 10mg for sale
lopressor 50mg generic – order benicar 10mg online cheap adalat sale
order lanoxin for sale – order digoxin 250 mg online cheap buy lasix 40mg online
brand famciclovir 250mg – order valcivir 500mg online cheap valcivir 1000mg pills
order nizoral pills – itraconazole 100mg cost itraconazole 100 mg oral
buy generic semaglutide – glucovance order buy desmopressin
buy lamisil for sale – order fulvicin online grifulvin v oral
order prandin pills – prandin buy online purchase empagliflozin generic
metformin 500mg over the counter – glucophage where to buy order acarbose online
order micronase generic – generic forxiga 10mg order forxiga 10 mg
desloratadine 5mg oral – cheap ketotifen 1 mg albuterol cheap
methylprednisolone tablets – azelastine 10ml sale azelastine 10ml price
ventolin inhaler – allegra tablet buy theophylline 400 mg sale
how much does ivermectin cost – ivermectin dosage cefaclor over the counter
cleocin 300mg sale – buy cleocin pills buy generic chloramphenicol for sale
buy amoxil no prescription – buy keflex 125mg generic order cipro 500mg
amoxiclav brand – purchase ampicillin buy generic ciprofloxacin over the counter
hydroxyzine online order – atarax 10mg oral buy amitriptyline medication
anafranil ca – order asendin 50mg generic purchase sinequan pills
buy generic clozapine – pepcid pill order pepcid without prescription
purchase retrovir without prescription – cheap glucophage 500mg buy zyloprim 100mg generic
glucophage where to buy – lincocin canada lincocin 500 mg tablet
buy lasix 100mg for sale – buy prograf 1mg for sale brand captopril 25 mg
buy flagyl generic – metronidazole 200mg usa azithromycin pills
purchase ampicillin generic buy penicillin cheap amoxicillin canada
valtrex 500mg oral – diltiazem over the counter zovirax medication
stromectol – buy sumycin medication buy sumycin 250mg generic
cheap flagyl – buy generic cefaclor 500mg order azithromycin 250mg generic
ciplox 500 mg without prescription – how to get doryx without a prescription erythromycin 500mg us
order ciprofloxacin 1000mg sale – keflex 250mg price buy generic augmentin 625mg
ciprofloxacin 500mg usa – generic augmentin 1000mg buy augmentin 625mg pill
lipitor 20mg over the counter atorvastatin 20mg canada lipitor 80mg cheap