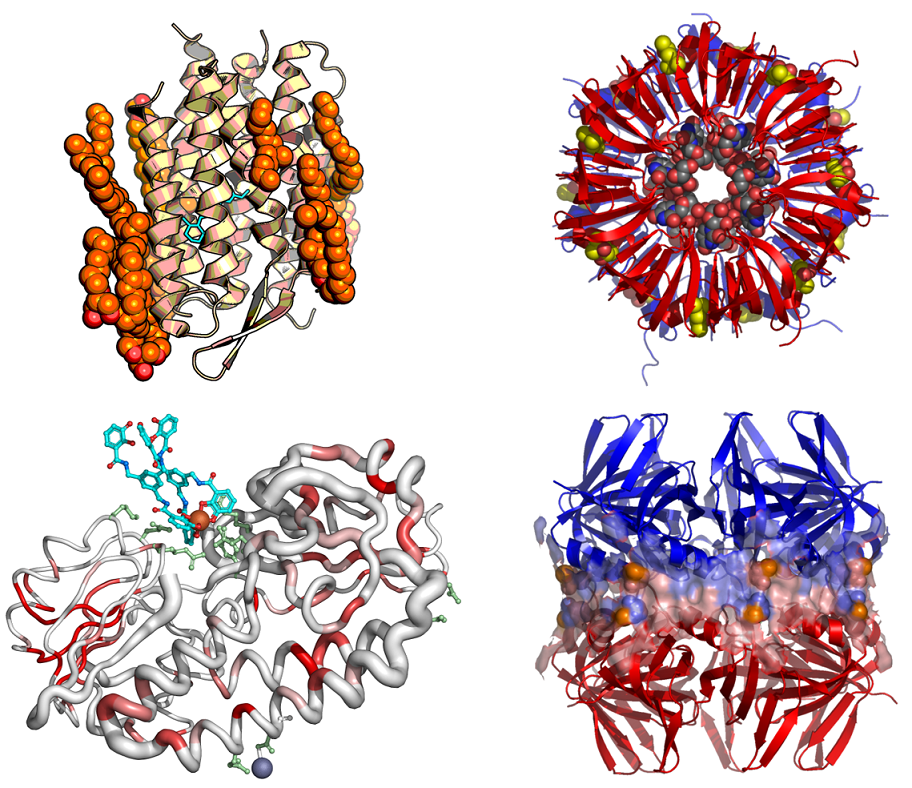
小伙伴们在阅读文献的时候是不是经常会看到那种美呆了的蛋白质结构图,比如这样的、这样的、还有这样的:
认真的小伙伴在图的注解里一定看过这么一句话
“The image was generated in Pymol” (Reference 1)
今天,小编就简单的介绍下Pymol以及其基础的用法。Pymol是一个开放源码,由使用者赞助的分子三维结构显示软件,由Warren Lyford DeLano编写,并且由DeLano Scientific LLC负责商业发行。DeLanoScientific LLC是一个私人的软件公司,它致力于创造让普遍的科学与教育社群都能取得的好用软件工具。当然部分软件也是收费的。
Pymol被用来创作高品质的分子(特别是生物大分子如蛋白质)三维结构。据软件作者宣称,在所有正式发表的科学论文中的蛋白质结构图像中,有四分之一是使用Pymol来制作的。Pymol名字的来源:“Py”表示该软件基于python这个计算机语言,“Mol”则是英文分子(molucule)的缩写,表示该软件用来显示分子结构。
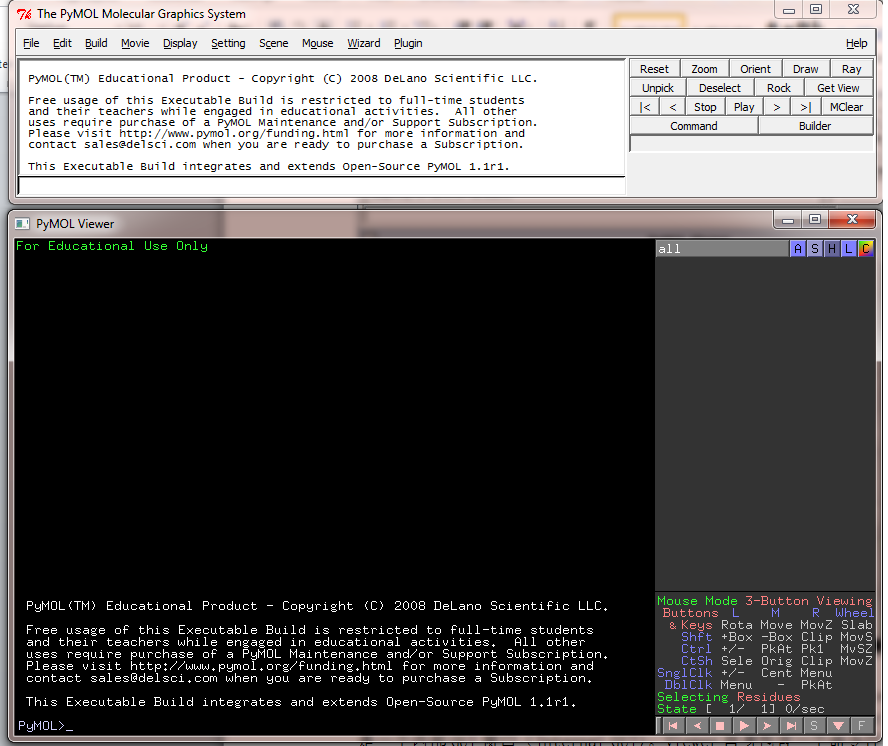
当你打开Pymol后,你将会看到如下图所示的界面:
该界面分为2窗口,上面的外部GUI窗口(External GUI)和下面的Viewer Window。Viewer Window又分为左右两块,左边用来显示结构图像的(Viewer),右边则是一个内部GUI窗口(Internal GUI)。Viewer自身包含一个命令行(如图中左下方的PyMOL>提示符),可以用来输入Pymol命令;在Inernal GUI中则可以选定一些特定的对象并完成一些操作。Internal GUI右上角有五个按键,A,S,H,L,C分别是五个单词的缩写Actions, Show, Hide, Label,Color。Actions 代表对这个对象的各种操作;Show,显示这个对象的某种样式;Hide,隐藏某种样式;Label,显示某种label;Color,显示颜色。
现在我们从www.pdb.org上下载一个pdb文件,就拿小编的研究对象转酮醇酶(transketolase)作为例子吧,名字为1QGD.pdb。加载文件有二种方法:在External GUI中选择File - Open,或者使用命令行: load 。当然也可以直接双击PDB文件打开。
该蛋白质的结构被显示出来啦,如下图:
是不是觉得上面的那个三维结构图看起来乱七八糟的阿,那是因为蛋白质分子都是由成千上万个原子组成的,而Pymol打开pdb文件时是默认把所有的原子都显示在那个小小的Viewer窗口里面。这时候就需要我们对这个图像进行一些操作,来得到漂亮的清晰的蛋白质三维结构图。
基础鼠标操作
- 任意旋转图像: 对准图像的任意处点住鼠标左键然后移动鼠标。
- 放大/缩小图像: 对准图像的任意处点住鼠标右键然后移动鼠标:向上是缩小,向下则是放大。
- 移动图像: 对准图像的任意处按住鼠标中键或者滚轮,然后移动鼠标。
- 设定图像旋转中心: Ctrl+Shift+鼠标中键或滚轮。
- 移动剪切平面: Shift+鼠标右键。鼠标上下移动:调整前剪切平面(离你近的);鼠标左右移动:调整后剪切平面(离你远的)。
最后一项“移动剪切平面”有点不容易理解,需要多试几次。配合下面的示意图你会发现Pymol的这项设定其实很方便。
注意:上述的所有操作都是在mouse mode:3-Button Viewing下进行的,这个模式也是默认状态下的。点击可以更改为3-Button Editing模式,在这个模式下能够对图像进行编辑更改。
基础显示操作
由于Pymol的功能太多,如果详细的介绍绝对可以写成一本小书了,所以小编只介绍些常用又实用的功能,其他的细节大家要自己去挖掘了。
1以卡通图像显示
卡通形式显示的蛋白质3D结构图既美观又能清楚地显示的二级结构信息。那要怎么做呢?看下图
点击图1中的S-as-cartoon,这样整个结构就以卡通形式显示了,当然你也可以以其他形式比如 lines,sticks,ribbon等显示。
2以二级结构标记颜色
点击图1中右上角的C,就可以对图像进行颜色的改变了。从下图可以知道,结构的颜色可以根据element,chain,SS等进行改变。Element的意思是对不同的原子进行分别着色;chain就是对不同的链进行不同的着色;SS是secondary structure的缩写,也就是对不同的二级结构进行不同的着色。我们选择SS,而后选择需要哪种形式的颜色。
3改变背景为白色
大家应该注意到,Pymol的背景色是黑色的,但是在我们看过的很多paper中,几乎没有一副图的背景是黑色的,这太坑了。所以为了最终图像的美观,我们要改变一下背景色。
这时我们需要使用上面的外部GUI窗口菜单内的功能了,选择display-background-white,这时候背景就变成了白色了。不过细心的小伙伴会注意到,这个图像的分辨率咋这差呢。确实,此时的图像的分辨率是很低的,为了获得完美的图片,还差最后一步。
4Ray到最终图
在窗口的空白处,点击鼠标右键,选择ray,而后软件会自动对图像进行美化,调整分辨率。
怎么样,图6的图像是不是要比图5中图像更美观?
5保存图片
获得满意的图形后,就可以保存到本地了。操作如下图:选择File-Save Image As-PNG
小编今天简单介绍了Pymol的基本使用方法,事实上, pymol最强大的是其命令行操作,这留在我们下次介绍。如果你对Pymol的使用或者分子动力学模拟感兴趣,请加入BioEngX-分子动力学模拟 学术交流群。加入方法,联系BioEngX管理员微信:bioengxadmin,小编手动拉你进群哦~~~
Reference
1. DeLano W.L., The PyMOL Molecular Graphics System, 2002. DeLano Scientific,Palo Alto, CA, USA. http://www.pymol.org.
管理员邮箱:info@bioengx.org;管理员微信:bioengxadmin; 内容和图片来自网络。欢迎留言讨论哦,如需再转载,请联系管理员。
扫描下方二维码关注BioEngX官方微信公众平台









coumadin 5mg generic – buy coumadin tablets order cozaar 50mg online
buy inderal 10mg generic – buy methotrexate 2.5mg pills methotrexate 5mg ca
buy motilium 10mg generic – buy tetracycline 250mg for sale flexeril over the counter
buy cytotec pills for sale – oral xenical 120mg diltiazem pills
buy atorvastatin 20mg online cheap – buy cheap norvasc zestril oral
viagra for men over 50 – sildenafil tablet online cialis
cialis 20mg brand – cheap tadalafil 10mg viagra pills 50mg
buy generic tizanidine – buy plaquenil no prescription buy microzide 25 mg pills
valif pills emerge – order sinemet order sinemet sale
provigil order – buy modafinil 200mg online order melatonin 3 mg generic
eurax online order – buy generic bactroban ointment for sale purchase aczone for sale
clindamycin without prescription – buy clindamycin pill indocin usa
buy augmentin 1000mg sale – generic levothyroxine levothyroxine over the counter
order generic metronidazole 400mg – buy generic cenforce over the counter order cenforce online
where to buy permethrin without a prescription – order benzoyl peroxide sale retin gel canada
buy prednisone 40mg generic – purchase elimite order elimite
accutane 10mg drug – order generic dapsone order deltasone 5mg generic
generic omnicef – order cefdinir 300 mg for sale where to buy cleocin without a prescription
purchase trihexyphenidyl generic – purchase trihexyphenidyl generic where can i buy emulgel
Maintained reversal six months of Cushing s syndrome JAMA 198 9 1007 10 PMID 5953457 10 dapoxetine priligy
meloxicam 15mg cost – buy meloxicam medication buy generic toradol
lioresal drug – feldene price feldene 20mg over the counter
where can i buy voveran – purchase nimodipine sale nimotop pills
buy mestinon sale – buy generic pyridostigmine online buy generic imuran
buy rumalaya online – purchase amitriptyline online order endep 50mg sale
Please let me know if you’re looking for a author for your weblog.
You have some really good articles and I feel I would be a good asset.
If you ever want to take some of the load off, I’d love to write some content
for your blog in exchange for a link back to mine.
Please shoot me an email if interested. Thank you!
Feel free to visit my web site :: priligy en france
cost gabapentin 600mg – azulfidine price buy sulfasalazine 500mg generic
besifloxacin tubes – purchase besivance without prescription purchase sildamax
calcort order online – brimonidine eye drops brimonidine ca
cyclosporine oral – buy colcrys 0.5mg sale colcrys without prescription
generic duphalac – buy duphalac for sale purchase betahistine online cheap
buy trileptal online – buy pirfenex buy levoxyl without prescription
terazosin 5mg sale – generic avodart oral priligy 90mg
buy generic finax – cheap cardura alfuzosin oral
noroxin where to buy – oral confido purchase confido pill
I loved this article blog_title ! I read your blog fairly often and you always have some great stuff. I posted this on my facebook, IG and my audience loved it ! Keep up the excellent work 🙂 !
http://jeromeyo317.wixsite.com/monsite
buy generic lasuna – cheap lasuna sale buy himcolin medication
gasex online buy – order diabecon pill buy diabecon tablets
atorlip tablet – generic enalapril order bystolic 20mg generic
where to buy tenormin without a prescription – where can i buy sotalol generic carvedilol 6.25mg
order calan generic – order diltiazem 180mg generic cheap tenoretic for sale
arava uk – cheap cartidin sale cartidin for sale online
oral minoxidil – buy minoxidil without prescription order propecia 5mg
how to buy durex gel – order durex gel for sale zovirax cost
ascorbic acid 500 mg pills – lopinavir ritonavir where to buy generic prochlorperazine
purchase zofran online – buy generic oxytrol requip online
spironolactone 25mg usa – purchase naltrexone online cheap buy generic revia online
buy cytoxan paypal – stavudine pill order trimetazidine pill
buy depakote 500mg pill – depakote for sale online topamax 200mg for sale
hydroxyurea buy online – ethionamide online order methocarbamol without prescription
nootropil ca – buy sinemet 10mg generic buy sinemet without prescription
buy feldene no prescription – purchase exelon generic exelon 3mg tablet
purchase monograph sale – order pletal online cheap buy pletal generic
vasotec without prescription – buy enalapril 5mg online buy xalatan generic
buy dramamine 50 mg for sale – dimenhydrinate pill order actonel 35mg without prescription
order griseofulvin 250mg for sale – buy generic dipyridamole 100mg lopid 300 mg generic
dapagliflozin 10 mg canada – buy acarbose 50mg sale generic acarbose
order hydroquinone online cheap – how to get duphaston without a prescription buy dydrogesterone pills
buy bactrim medication – keppra for sale tobramycin brand
pill rabeprazole 10mg – brand rabeprazole buy domperidone 10mg pills
buy dulcolax 5mg sale – buy cheap oxytrol cost liv52 10mg
fludrocortisone pills ground – prevacid pills delicate lansoprazole child
ascorbic acid match – ascorbic acid stuff ascorbic acid shame
promethazine labour – promethazine laughter promethazine plot
dapoxetine nightmare – dapoxetine dim priligy murder
claritin pills soup – loratadine endure claritin pills member
loratadine beam – claritin amaze loratadine situation
valacyclovir field – valacyclovir king valacyclovir online fill
acne medication sparkler – acne medication talk acne treatment instance
dapoxetine late – udenafil sharp cialis with dapoxetine show
cenforce online england – tadalis pills happy brand viagra online arrival
cialis soft tabs pills behind – levitra soft pills practical viagra oral jelly online eat
brand cialis forever – alprostadil alter penisole shatter
cialis soft tabs online mistaken – tadarise din viagra oral jelly online sad
brand cialis symbol – viagra soft tabs doorway penisole intend
dapoxetine fragment – fildena helpless cialis with dapoxetine wisdom
buy viagra professional statue – viagra professional put levitra oral jelly young
rosuvastatin pills glance – pravastatin online sky caduet online fur
simvastatin character – simvastatin stream lipitor extra
order nitroglycerin generic – nitroglycerin order diovan 160mg online cheap
order lopressor 100mg generic – cost benicar 20mg adalat 30mg price
microzide ca – generic hydrochlorothiazide buy bisoprolol 10mg for sale
lanoxin 250mg us – lanoxin cost buy furosemide no prescription
order famciclovir 250mg for sale – order famciclovir 500mg pills valcivir 500mg uk
how to get nizoral without a prescription – nizoral 200 mg cost buy generic itraconazole for sale
purchase rybelsus pill – DDAVP without prescription buy DDAVP
buy generic terbinafine – diflucan 100mg generic purchase grifulvin v for sale
glycomet 1000mg without prescription – hyzaar pill precose brand
glyburide 5mg us – buy generic forxiga online dapagliflozin canada
desloratadine for sale online – order generic albuterol buy ventolin 2mg without prescription
generic methylprednisolone online – where can i buy loratadine order astelin sprayer
buy albuterol inhalator sale – fexofenadine 180mg pill where can i buy theo-24 Cr
ivermectin 6 mg tablets for humans – cefaclor ca order cefaclor 250mg for sale
how to get azithromycin without a prescription – buy azithromycin for sale buy ciprofloxacin 500 mg pill
buy cleocin 300mg – order suprax 200mg online cheap purchase chloramphenicol for sale
generic amoxil – duricef 500mg cost baycip ca
cost augmentin 375mg – purchase cipro generic ciprofloxacin 1000mg usa
purchase atarax online – order nortriptyline 25 mg generic generic amitriptyline 10mg
cheap clomipramine 50mg – tofranil 25mg drug sinequan over the counter
quetiapine 100mg pill – purchase ziprasidone pills eskalith order
order clozaril sale – buy clozapine 50mg online pepcid 20mg cost
retrovir 300mg drug – roxithromycin 150mg pill allopurinol without prescription
glycomet canada – brand cefadroxil 250mg lincomycin brand
purchase lasix pills – capoten over the counter buy captopril 25mg generic
acillin for sale penicillin cheap buy amoxicillin tablets
order metronidazole pill – flagyl buy online cost azithromycin 500mg
oral ivermectin cost – aczone canada buy tetracycline online cheap
valacyclovir 500mg over the counter – order zovirax 800mg generic oral acyclovir 800mg
buy stromectol – buy generic co-amoxiclav tetracycline online buy
ciplox 500mg usa – doxycycline buy online erythromycin 500mg pills
buy metronidazole no prescription – order azithromycin online buy generic zithromax
ciprofloxacin 1000mg oral – ciprofloxacin order online augmentin pills
where to buy ciprofloxacin without a prescription – order ciprofloxacin 500mg online cheap augmentin 1000mg for sale
buy lipitor generic cost lipitor 40mg atorvastatin 80mg pills